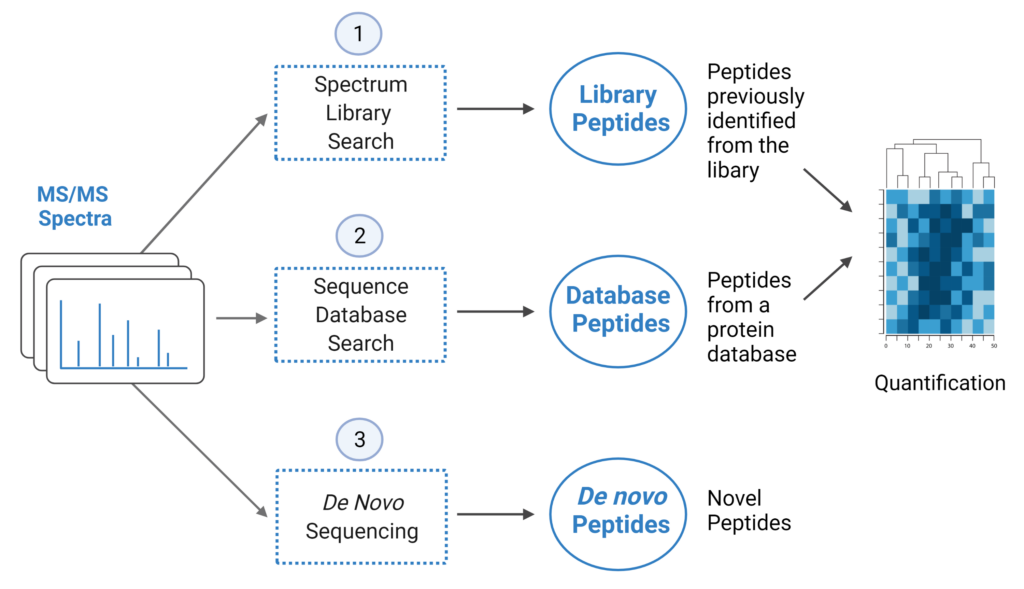
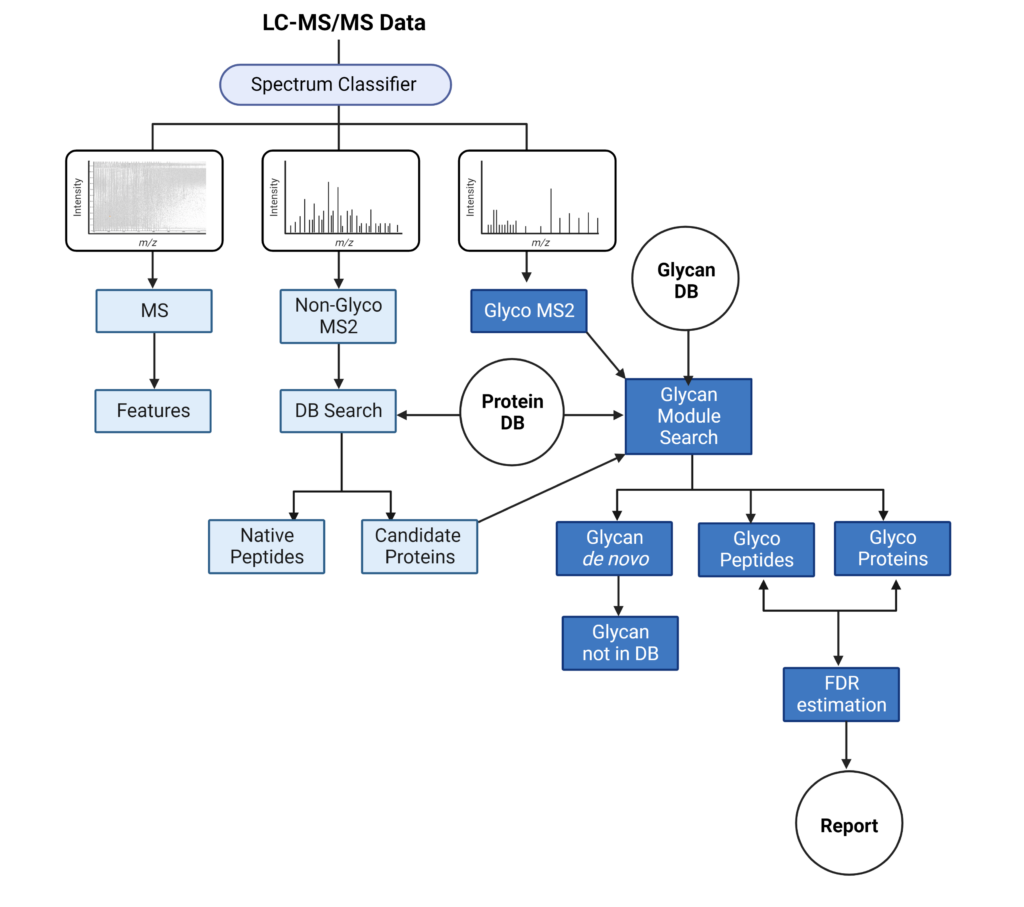
蛋白质组学是研究特定类型细胞、组织或生物体在特定时间、特定生物条件下表达的蛋白质的科学。自下而上的蛋白质组学是在将蛋白质酶切为多肽片段,然后通过质谱检测的最常用的方法。
PEAKS® Studio 是一款由深度学习技术驱动的蛋白质组学专业软件,可以满足发现蛋白组和靶向验证蛋白组数据分析需求。完全重新设计的架构,能够提供更高的速度和稳定性。通过更新的图形用户界面,获得PEAKS®直观的数据可视化,在全新的外观和优化的工作流程中,简化用户的数据分析。PEAKS® Studio可以实现多肽/蛋白质定性定量、翻译后修饰位点和突变位点鉴定等功能,提供了蛋白质组学数据分析的完整解决方案,将您的研究提升到新的高度!
PEAKS Studio 12
仪器商中立,支持DDA和DIA数据分析的完整软件方案
PEAKS® Studio 12 提供完整的自下而上的蛋白质组学解决方案,具有更高的准确性、灵敏度和速度。原有工作流均进行了中大升级,新版本的DeepNovo Peptidome工作流使得PEAKS®成为非经典多肽和蛋白质鉴定的唯一选择。
软件功能亮点
- 升级后的DeepNovo 可通过FDR对从头测序肽段进行质控
- 利用深度学习进行从头测序辅助的数据库搜索,以最大限度提高肽段鉴定效率
- DIA 工作流程(谱图库搜索 + 直接数据库搜索 + DIA de novo测序)
- 提高直接数据库搜索的灵敏度和准确性。
- 基于深度学习的技术,增强谱图、保留时间和碰撞截面值预测。
- Hybrid-DIA支持靶向和全局蛋白综合分析
- 包含 500 多种修饰的翻译后修饰 (PTM)信息
- 序列变异和突变位点鉴定
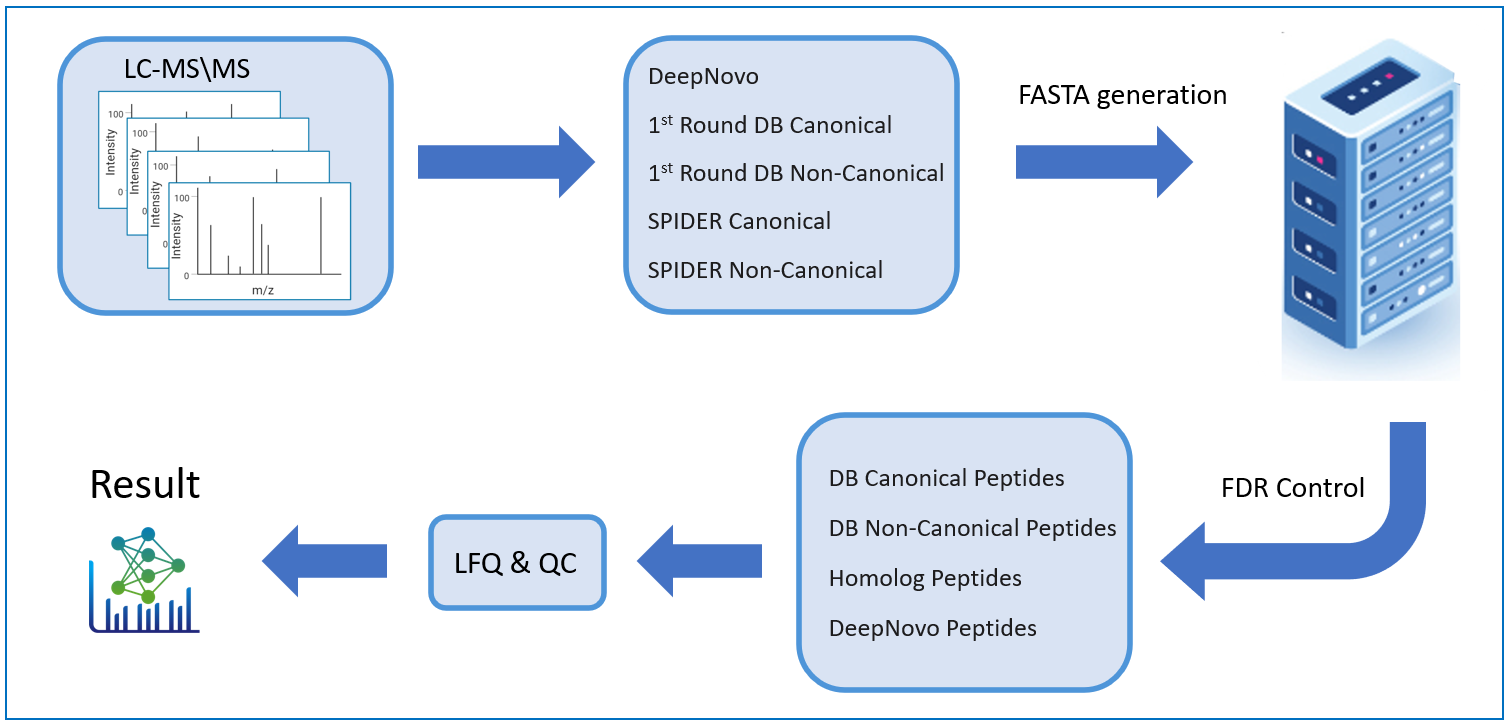
- DeepNovo 免疫肽组学工作流:集成经典和非经典数据库搜索、同源性搜索和 DeepNovo,通过 FDR质控提升HLA多肽鉴定
- DIA 多肽组工作流
- 非标记和标记定量:TMT (MS2, MS3) / iTRAQ,SILAC, 18D标记,ICAT,以及用户自定义的定量方法等。
- 定量结果可视化:PCA,热图,火山图、相关性分析、提取离子色谱图(XIC)等
- 易于使用的图形用户界面(GUI)可以查看和展示数据的细节信息,方便用户过滤和验证结果。
- 谱图库查看工具使用户可以在使用前评估质量和验证谱图库。
- 以可视化的方式展示统计结果,以评估原始数据和结果的质量。
- LC-MS/MS 热图提供多肽特征的完整可视化,有鉴定信息的MS/MS谱图所处的位置,并与之相关的质荷比(m/z),保留时间(RT),补偿电压(CV),离子淌度(1/k0)和信号强度等
- PEAKS IMS 附加模块:支持离子淌度质谱蛋白质组学(timsTOF Pro、FAIMS)。
- 针对每种仪器和碎片类型进行专门优化,以确保最佳的准确性和灵敏度。
- 全面支持DDA和DIA的定性定量分析。
软件概览
算法
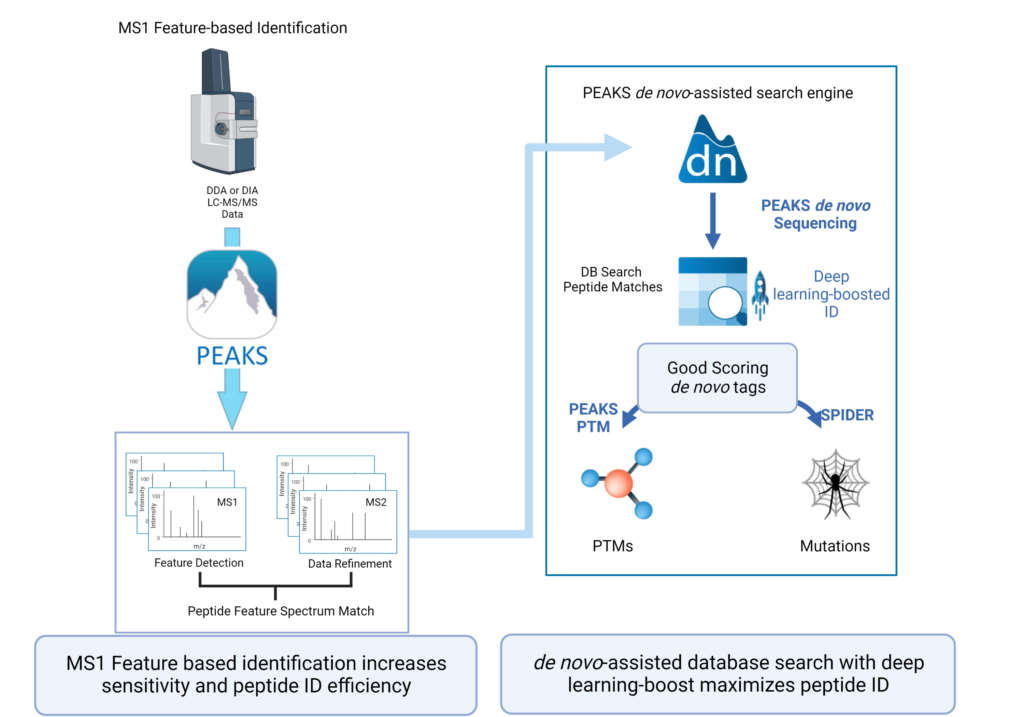
PEAKS 12使用最新的PEAKS算法进行所有分析,包括数据优化、定性和定量。用户在PEAKS Studio 12中可以选择使用深度学习增强的ID工作流进行DDA分析,鉴定率可提高10%以上。
整合直接序列数据库搜索的一体化DIA工作流
PEAKS Studio 12 提供独特的DIA 工作流 通过整合三种方法最大限度地鉴定多肽:谱图库搜索、直接数据库搜索和从头测序,定性定量提升高达 20%。
- SL search是针对于预定义的谱图库进行的。不包含在谱图库中的多肽可以用direct DB的方法直接对蛋白的序列数据库继续搜索
- DIA数据可以直接对蛋白序列数据库进行搜索。优化的机器学习算法可以提高多肽鉴定的灵敏度与准确度
- 在序列数据库中未得到好的多肽匹配的谱图,可以进行从头测序
- 无论是来自于谱图库搜索还是直接序列数据库搜索的多肽鉴定结果,都可以进入到下一步的定量分析
注意:在PEAKS中,以上三种方法:PEAKS SL search,Direct DB search 和从头测序都是可选的,并且可以按顺序组合或者单独执行分析,对于用户不同的分析目的,工作流的定义更为灵活。
基于特征峰的定性工作流提高了灵敏度,实现多肽鉴定最大化
- 专为DDA设计,提高了数据分析的重现性
- 整合了数据库搜索和从头测序,实现深度蛋白质组学
- PEAKS DDA的启用深度学习增强功能,使多肽的鉴定达到最大化
点击这里学习更多关于从头测序,助力您的蛋白质组学和多肽组学研究。
PEAKS DeepNovo 多肽组工作流:为免疫多肽设计的优化解决方案
该工作流是专门为多肽组学研究设计的,结合了数据库搜索、从头测序和多肽突变的鉴定。 Learn more
离子淌度定量分析,最大程度减少缺失值并提高准确性
PEAKS Glycan 分析模块:确定糖基化位点和糖结构信息
PEAKS Glycan 糖分析模块可以分析复杂的糖蛋白质组学数据,并为用户提供高灵敏度和可靠性的结果,帮助用户更深入的了解样品中糖蛋白质组的信息。Learn more
- 可以通过PEAKS Glycan全面了解糖基化肽段和糖蛋白
- 对糖蛋白组深度表征
- 创新型糖肽的鉴定软件
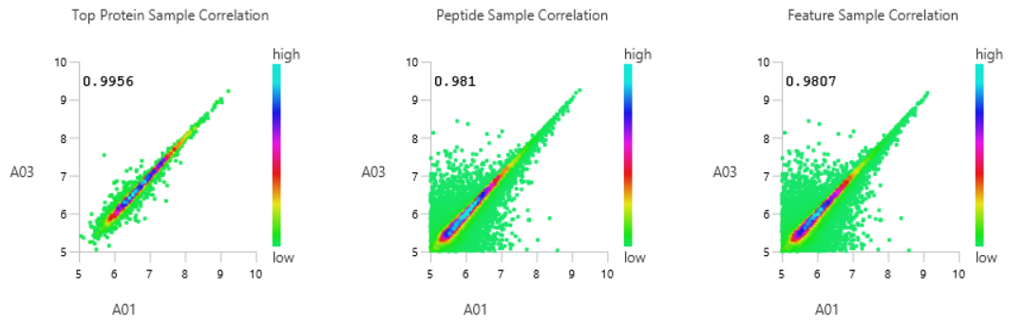
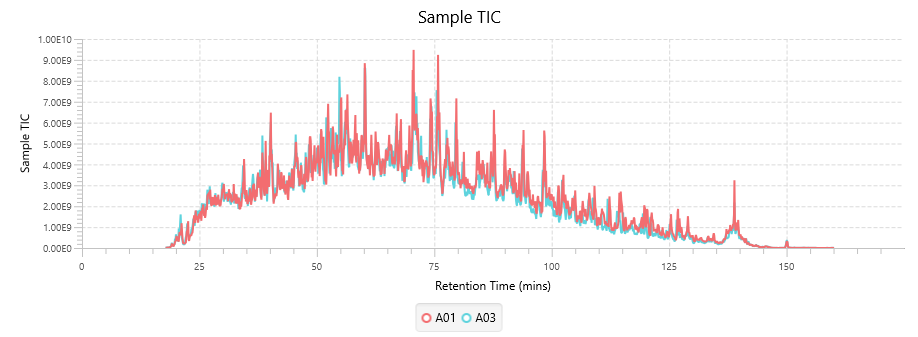
质量控制(QC)功能——从原始数据到结果可视化
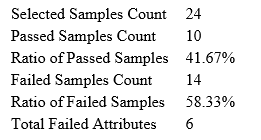
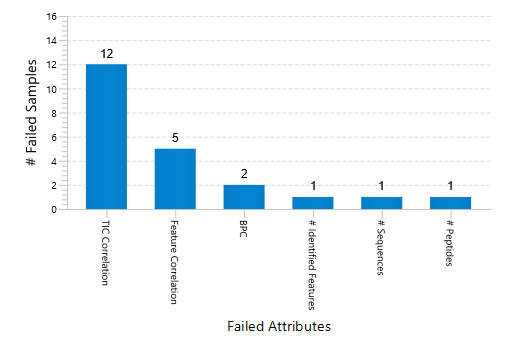
使用PEAKS®Studio 12中的全新质量控制(QC)分析,用户可以评估原始数据和结果的统计信息,并获得LC-MS采集的关键指标。这个自动化工具是为DDA和DIA数据设计的,新版本的Peptidome工作流也已嵌入,便于快速筛选异常数据。
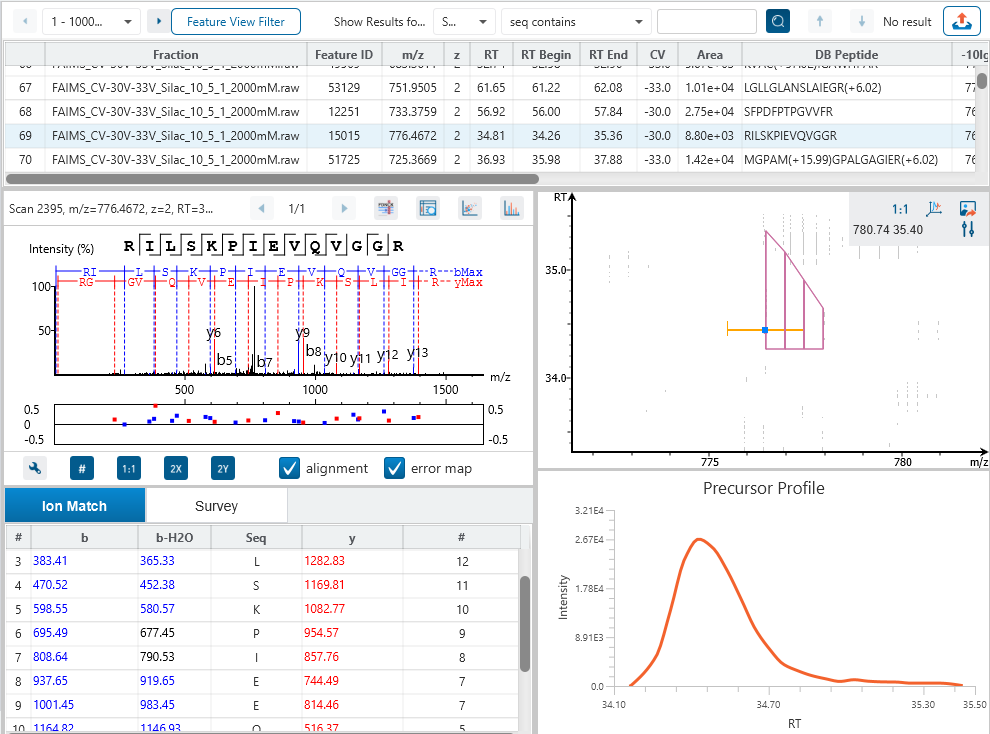
多肽特征峰视图
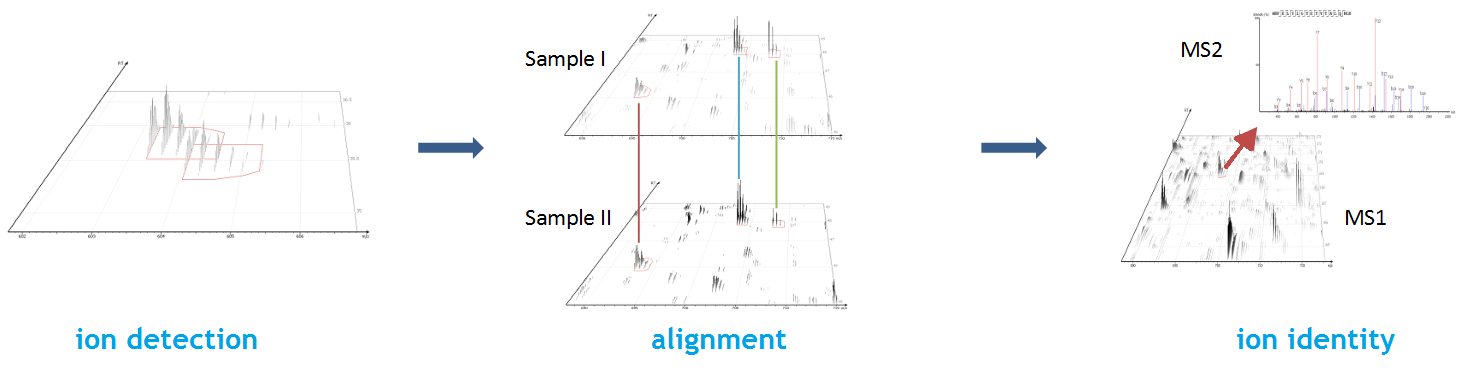
从data refinement到定性、定量,PEAKS 12是基于多肽特征峰进行分析的。多肽特征包括一系列对应的m/z值、保留时间范围和不同的同位素峰形成的信号强度,以及离子淌度维度的信息。
In PEAKS identification results, users will find a “Feature” tab which presents all details of every peptide feature in the raw data, visually and tabularly. In the Peptide Feature table, PEAKS conveniently summarises feature details detected from LC-MS or LC-IMS/MS, including the identified sequence as determined by database search, DDA, or de novo sequencing. Each feature is also linked to their corresponding protein view, spectrum view and LC-MS or LC-IMS/MS view for further inspection.
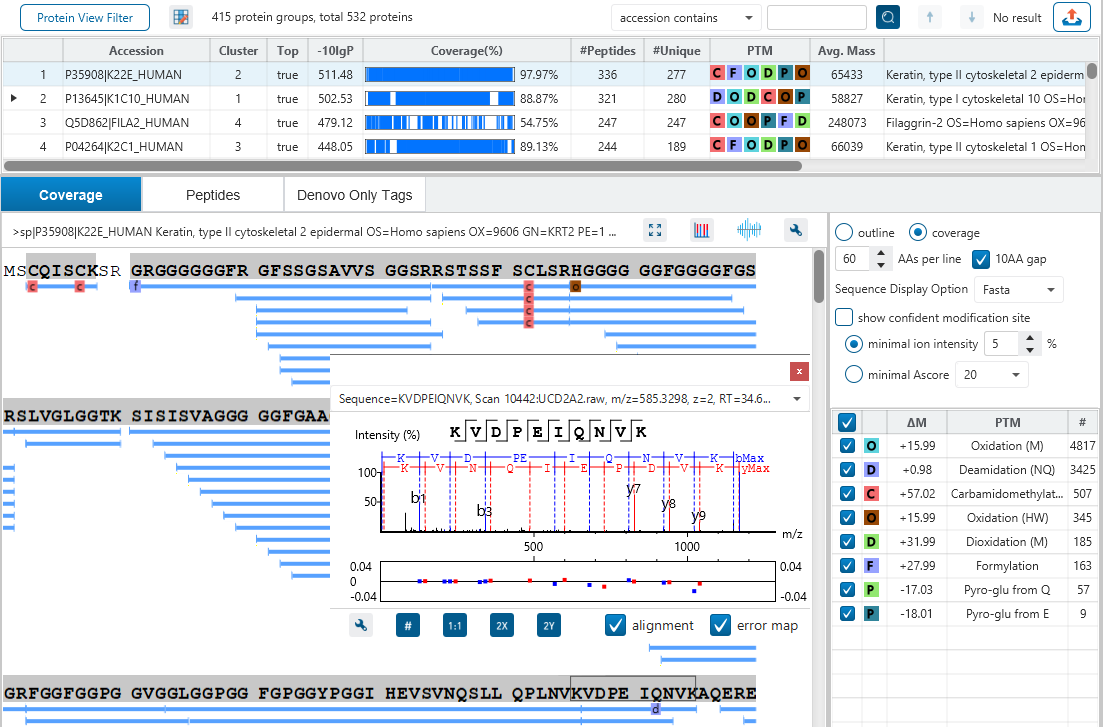
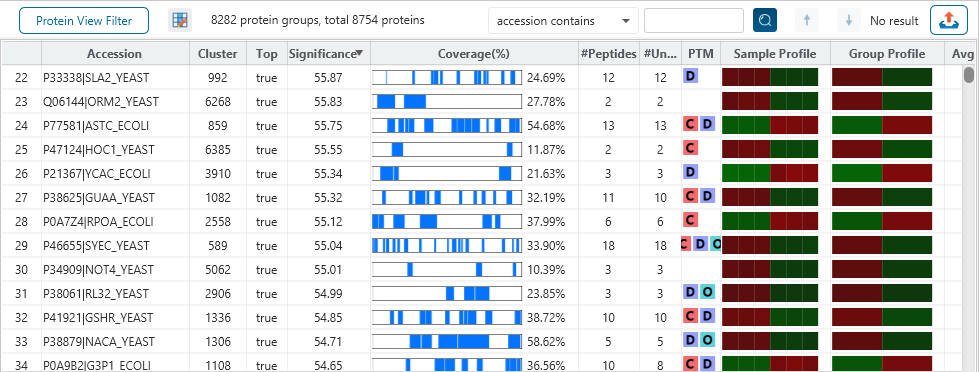
The Protein View
The Protein View presents protein profiling across complex biological samples in both DDA and DIA workflows. For each protein, the Sequence Coverage View displays a peptide map with spectrum annotation for validation. With PEAKS traditional de novo-assisted database search, users easily view identified peptide sequence in blue, while a grey bar indicates a de novo only tag match.
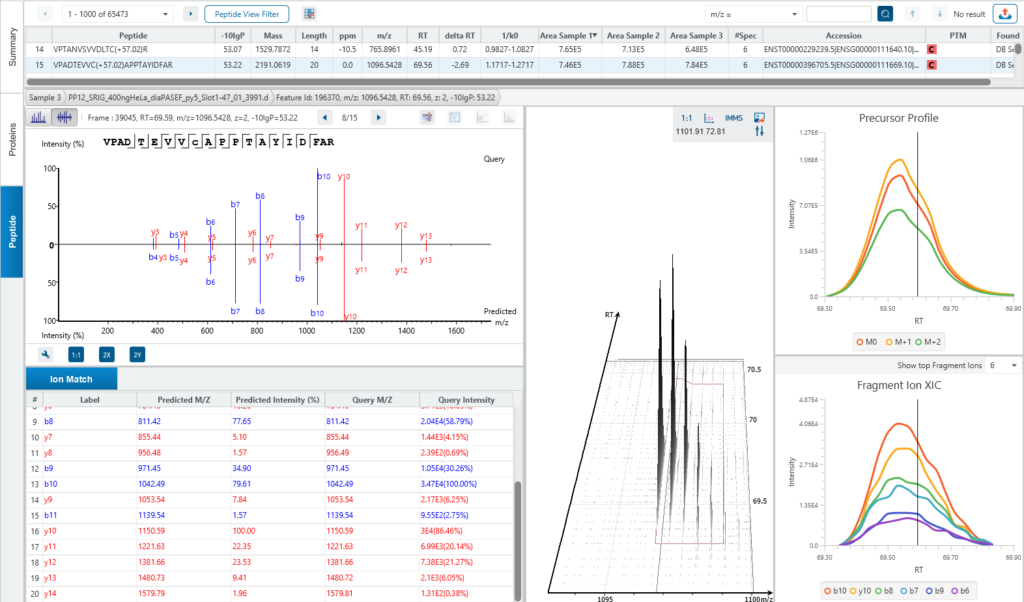
The Peptide View
The Peptide View provides a list of identified peptides with the abundance from MS1. For each modified peptide, the confidence of modification site (Ascore) is associated.
The Quantification View
With the add-on module of PEAKS Q, PEAKS Studio also determines relative protein abundance changes across a set of samples simultaneously and without the requirement for prior knowledge of the proteins involved.
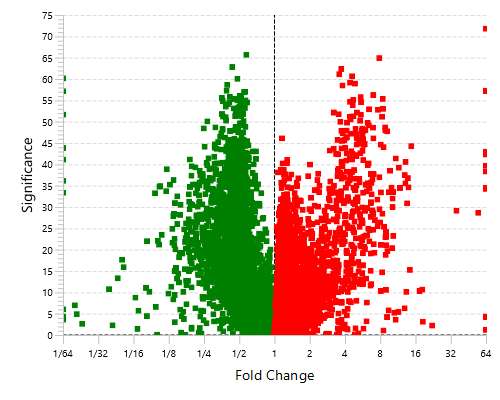
Highly differentially expressed proteins between two groups are identified by statistical analysis tool (fold change >2, FDR <0.01) and displayed in a heatmap format.
授权信息
PEAKS Studio 可以根据您实验室的使用需求选择授权版本
- Desktop – 24 线程, 最多可以处理达到CPU的24核心
- Workstation – 48 线程,最多可以处理达到CPU的48核心
硬件配置
PEAKS Studio 12建议安装在 Windows 10 或更高版本的 64 位 Windows 操作系统上。
推荐配置:
Desktop License: 30+ threads processors and 64 GB+ of RAM with compatible GPU (described below)
Desktop版本:30+ 线程处理器、64 GB+ RAM 以及兼容 GPU(如下所述)
Workstation版本:60+ 线程处理器、128 GB+ RAM 以及兼容 GPU(如下所述),如 Intel Core i7/i9/Xeon 或 AMD Ryzen 7/9/Threadripper 处理器
如果需要运行DeepNovo,要求机器配备NVIDIA CUDA计算能力≥8 且≥8GB内存的GPU。
运行 DIA 直接数据库搜索,建议机器配备 NVIDIA CUDA 计算能力≥5 且≥8GB 专用内存的GPU。
GPU 驱动必须更新到 CUDA 12.3 或更高版本
References & Resources
References
- Tran NH, Qiao R, Xin L, Chen X, Liu C, Zhang X, Shan B, Ghodsi A, Li M, Deep learning enables de novo peptide sequencing from data-independent-acquisition mass spectrometry, Nat. Methods, 16, 63–66 (2019). https://doi.org/10.1038/s41592-018-0260-3
- Tran NH, Zhang X, Xin L, Shan B, Li M. De novo peptide sequencing by deep learning. Proc. Natl. Acad. Sci. U.S.A. 114, 8247-8252 (2017). https://doi.org/10.1073/pnas.1705691114