High Throughput, Multi-User,
Proteomics LC-MS/MS Analysis
Software
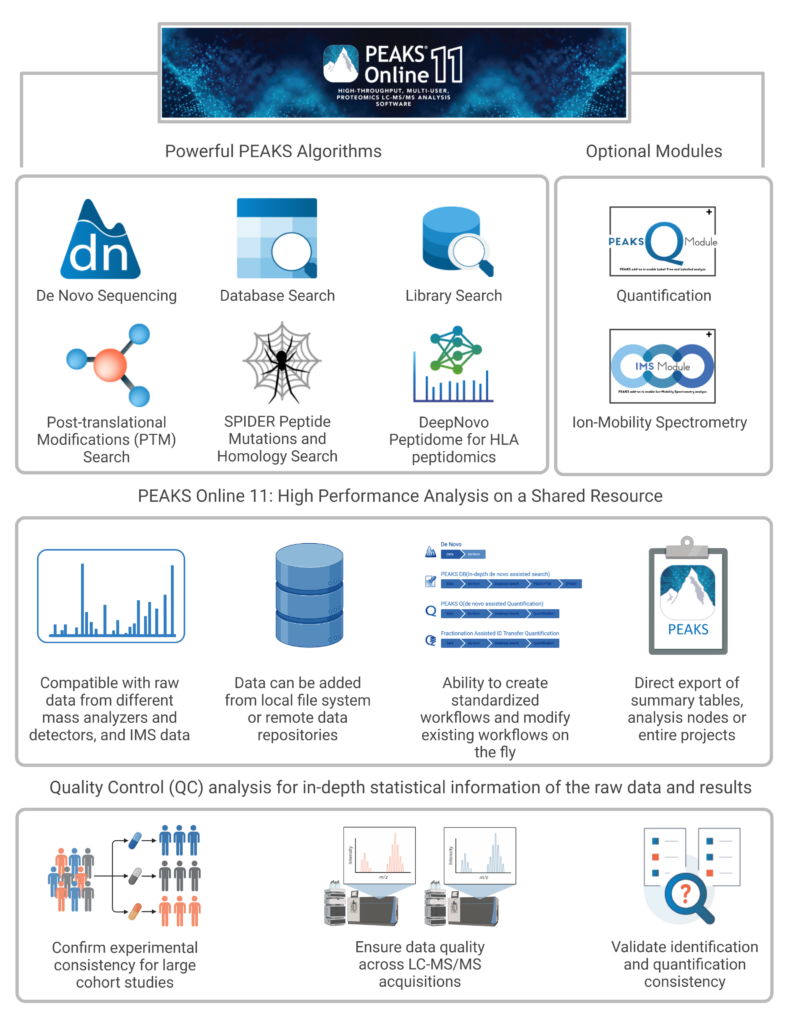
PEAKS Online 11
基于服务器的高通量蛋白质质谱数据全流程自动化解决方案
PEAKS® Online 11 是一款适应于蛋白质组学大数据时代,能够充分利用服务器计算资源,对大规模质谱数据集提供并行运算,同时可执行复杂算法得到精确结果的软件平台。PEAKS Online可以部署在任意集群、多 CPU服务器或者云服务器上,并且可以根据计算需求,在授权范围内,弹性扩展PEAKS Online所需的计算资源。
软件功能特点
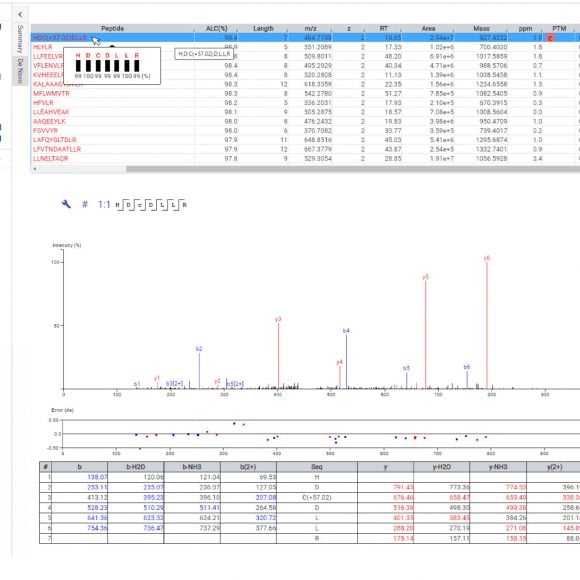
- 从头测序
- 通过全新的深度学习技术优化了数据库搜索,大大提高了鉴定的深度和效率
- DIA工作流(Spectral Library Search + direct DB + de novo)
- 新功能:直接序列库搜索优化,提高了灵敏度和准确度,支持DIA短梯度数据
- 通过谱图、保留时间和CCS值预测的深度学习算法,强化了DIA的数据分析
- 新功能:来自于DIA数据de novo结果的谱图库构建
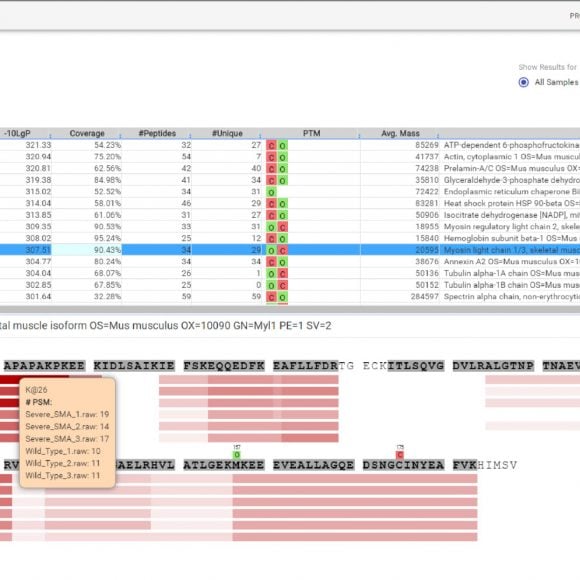
- PEAKS PTM可一次分析313种天然翻译后修饰
- 序列变异和突变分析
- 新功能:基于DeepNovo的多肽组工作流:整合de novo和同源搜索并提供了全局的FDR质控的方法,提升了多肽组的分析。
- TMT (MS2, MS3) / iTRAQ,SILAC, 18D标记,ICAT,以及用户自定义的定量方法等。
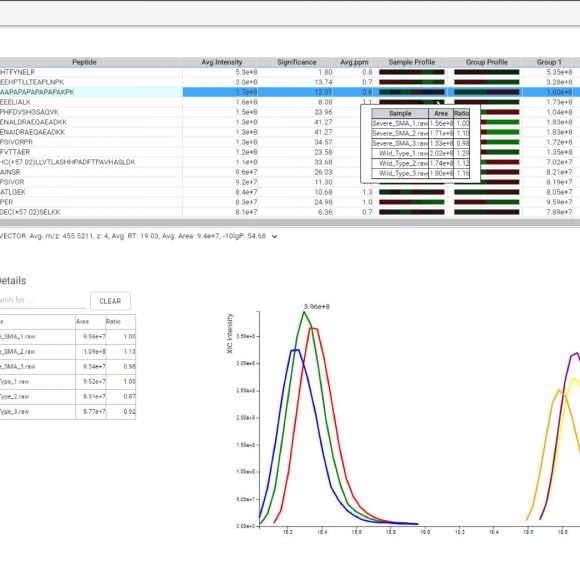
- 定量结果可视化:热图,火山图、相关性分析,提取离子色谱图等
- 新功能:QC(质量控制)-增强大队列研究(DDA和DIA):定性相关和定量相关的质控分析,以确保实验一致性,数据的质量,定性和定量结果的一致性。
- 详细和易于使用的图形用户界面(GUI)方便用户查看、过滤和验证结果。
- 以可视化的方式展示统计计算的结果,以评估原始数据和/或结果的质量。
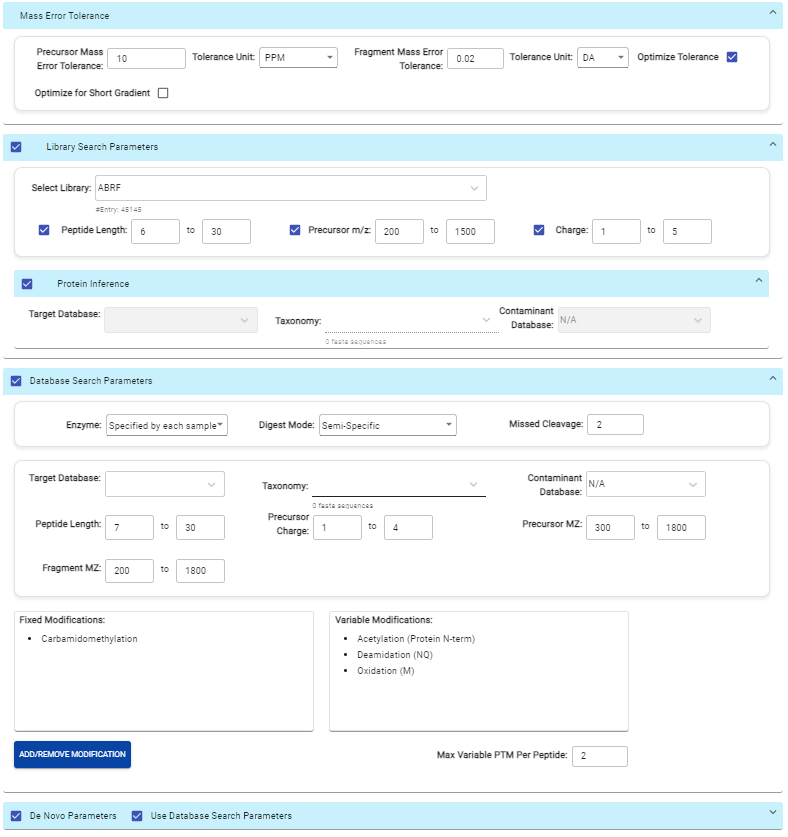
- 用户友好的界面,可以创建和保存工作流程,包括搜索参数和数据库,方便执行日常分析
- 使用PEAKS daemon实现真正的自动化:自动化连接仪器,可以在工作流中设置从数据采集到分析和结果产出的无缝衔接。
- 将团队协作与后台管理控制相结合,将标准化工作流、数据库、PTM、定量方法的设置以及项目,实现内部共享
- GPU和基于集群的解决方案,加速数据分析
- ZenoTOF
- 支持离子淌度蛋白质组学数据(timsTOF Pro,FAIMS,HDMSe)
- 算法针对每款仪器和碎片类型进行适配优化,以确保最佳的精度和灵敏度。
- 全面支持DDA和DIA数据的定性和定量分析
专为高通量分析需求设计的分布式计算系统
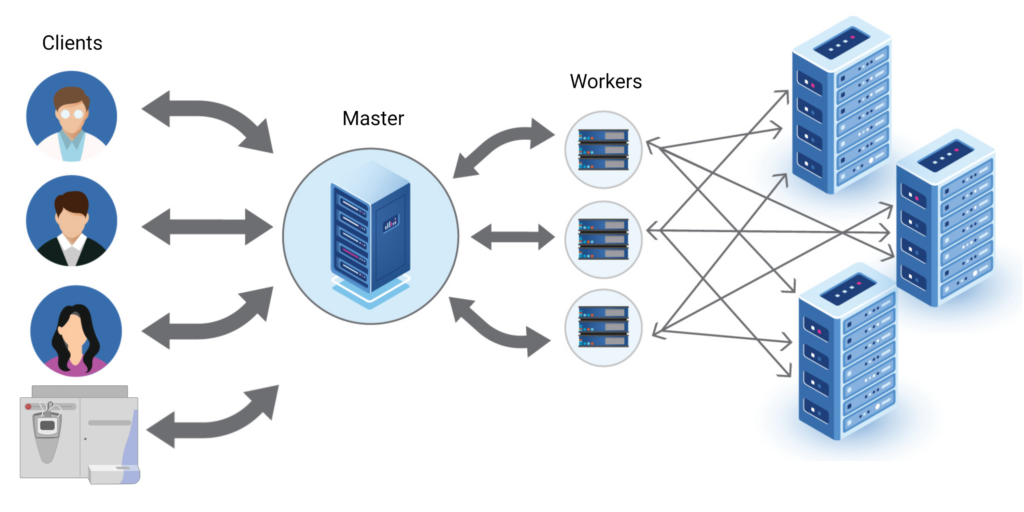
PEAKS Online 11采用最新的分布式计算技术,通过为网络中的多个用户建立PEAKS工作流来实现高通量性能。Master是PEAKS Online系统的中心,它接受来自客户端的计算请求或使用自动化系统直接来自MS仪器的计算请求;PEAKS daemon:实现自动的仪器连接。 Master将所有的计算负荷分配给不同的Worker,Worker即运行计算的部分(CPU和GPU)。所有计算所需的数据都存储在分布式数据库系统中。由于计算和数据存储都分布在许多不同的节点上,单个节点故障不会阻塞整个系统。因此,系统始终对用户是可用的。此外,由于分布式系统也具有高度的可扩展性,能够随时添加额外的节点以实现最大性能。
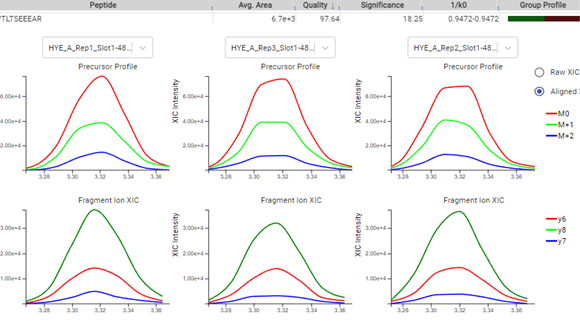
优化的DIA Workflow: Library Search, Direct Database Search, de novo sequencing
DIA 分析在近年越来越流行。在过去,DIA方法依赖于从DDA生成谱图库来对多肽进行定性和定量。
PEAKS Online 11提供了一个独特的DIA工作流程,通过集成三种方法,最大限度地实现多肽鉴定:谱图库搜索(SL search),直接数据库搜索(direct DB)和从头测序(de novo sequencing)。
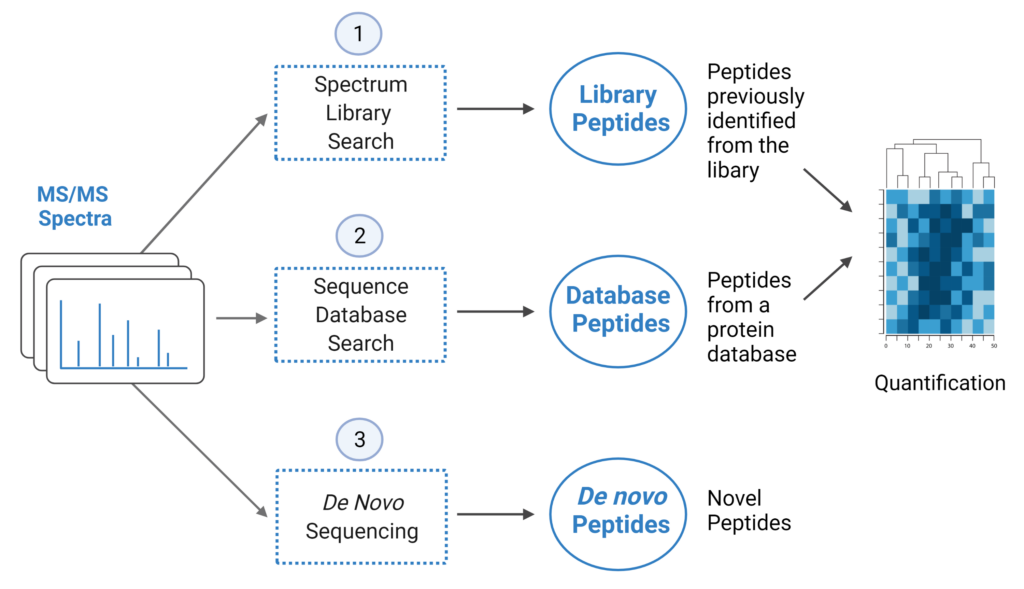
- SL search是针对于预定义的谱图库进行的。不包含在谱图库中的多肽可以用direct DB的方法直接对蛋白的序列数据库继续搜索
- DIA数据可以直接对蛋白序列数据库进行搜索。优化的机器学习算法可以提高多肽鉴定的灵敏度与准确度
- 在序列数据库中未得到好的多肽匹配的谱图,可以进行从头测序
- 无论是来自于谱图库搜索还是直接序列数据库搜索的多肽鉴定结果,都可以进入到下一步的定量分析
注:所有定性方法,PEAKS Spectral Library search, Direct Database search, de novo sequencing, 以及PEAKS Q, QC都是可选的,并且可以按顺序组合或者单独执行分析,对于用户不同的分析目的,工作流的定义更为灵活
选择谱图库搜索,然后直接数据库检索,可以提高蛋白质鉴定率,而de novo测序进一步提高了蛋白质组的完整性。除了深入的识别分析,PEAKS Online 11还允许通过LFQ对DIA数据进行量化。最后,可选的QC (Quality Control)将提供与鉴定和定量相关的质量控制措施,以确保实验的一致性、数据质量以及鉴定和定量的一致性。

Quality Control (QC) 功能实现原始数据和结果的深度剖析
通过PEAKS®Online 11中的新的质量控制(QC)分析,用户可以评估原始数据和/或结果的统计信息,并获得LC-MS采集属性的有益洞察。这个自动化工具是为DDA和DIA数据设计的,并将提供确定数据质量和评估实验设置的元素。
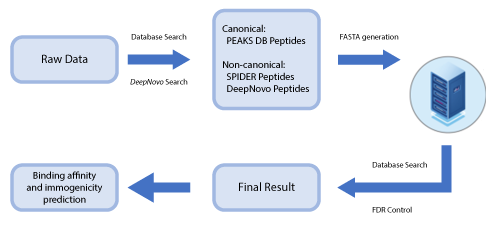
PEAKS DeepNovo 多肽组工作流: 为免疫多肽设计的优化解决方案
这个新开发的解决方案是专门针对多肽组学数据分析的工作流程,它结合了数据库搜索、从头测序和突变肽的鉴定。
数据非依赖采集(Data-independent acquisition, DIA)已经成为一种深度蛋白质组学鉴定的强大工具,PEAKS Online现在已经支持免疫肽组学的DIA数据分析。新的DeepNovo Peptidome已嵌入PEAKS Online的DIA工作流中,用于免疫肽组学分析,同时整合了谱图库检索、序列数据库搜索和从头测序。Learn more
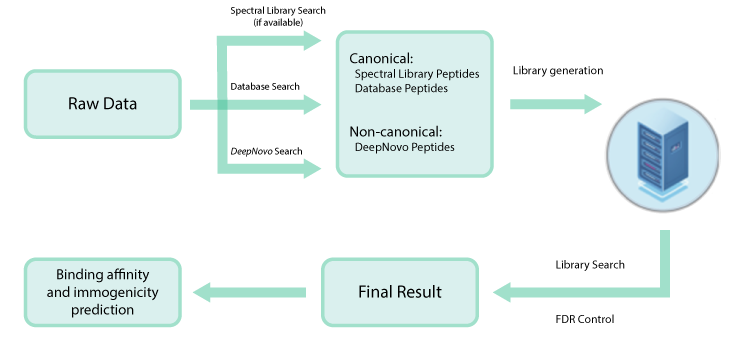
PEAKS Workflow
PEAKS Online 11旨在使用PEAKS 11软件工作流程进行准确而灵敏的蛋白质组学分析,通过共享计算资源,实现更高的分析性能。用户能够执行和PEAKS Studio中一样的 de novo Sequencing![]() , PEAKS DB Database Search
, PEAKS DB Database Search![]() , PEAKS Library Search
, PEAKS Library Search![]() , PEAKS PTM
, PEAKS PTM![]() , 和突变分析 SPIDER
, 和突变分析 SPIDER![]() 。可选模块PEAKS Q
。可选模块PEAKS Q![]() , and PEAKS IMS
, and PEAKS IMS![]() 可以用于蛋白质组相对定量和对离子淌度数据的支持。
可以用于蛋白质组相对定量和对离子淌度数据的支持。
PEAKS Online为用户提供了更有效和更大规模地利用既定PEAKS工作流的能力。我们把用户向服务器发送、递交数据分析的交互式工具称之为PEAKS的客户端,结果的展示类似于PEAKS Studio的展示方式。通过Web客户端界面或者命令行的客户端界面,可以接受多用户同时访问PEAKS Online 服务器,支持分析项目水平和数据水平的并行处理。
智能数据处理的需求,就是如何实现高效数据处理。如果需要使用一组新的参数重新运行分析,对于则不需要重新处理整个工作流。对于时相分析,以及样本可以陆续添加到现有的项目中,这种应用场景而言,PEAKS Online是完美的解决方案,工作流可以在联机状态下实时修改和添加。PEAKS Online 11将只处理需要更新的数据,同时保留以前生成的任何相关信息。一旦生成了所需的结果,用户可以导出每个样本的结果,或者一次导出所有结果。
在PEAKS Online的双客户端接口下,易于集成在任何的蛋白质组数据分析工作流中。Web客户端界面提供了一个可视化的交互界面,在此设置和提交项目,并查看和验证结果。命令行(CLI)客户端可集成在现有的工作流水线中,实现自动化地数据处理。

浏览器可视化客户端
访问管理工具
设置、查看、共享分析结果
监测分析进程,修改杈限
支持任意操作系统
在任意操作系统的计算机上都可以很容易
地访问PEAKS的分析结果
在PEAKS Online中,用户不需要安装其他
软件来访问服务器,只需通过Chrome浏览
器,设置参数,递交分析,查看结果。
用户可以设置让系统可以在结果分析完成
后发邮件提醒。
适用于普通用户执行分析,无需额外的计
算机技能。

命令行客户端
自动化数据批处理
定义工作流
易于整合到现有数据流程中
减少人工处理
命令行接口可以使用户对执行完全自动化
地数据分析。
轻松搭建标准化的工作流,以便于在数据
存储中创建数据
PEAKS Online x可以实现分析与产出结果
的同步化进行。
由命令行接口可以自动导出.csv格式的结
果文件,也可以通过web客户端查看。
PEAKS Online 使用分布式计算技术来充分利用硬件的计算能力。PEAKS Online 架构是建立在非常流行的Apache Cassandra数据库系统上的。PEAKS Online 使用Akka Actor system实现有效地超高性能的并行计算,特别适合实现高度自愈的容错系统。这种软件架构方式,在PEAKS Online中可以实现PEAKS Studio不能实现的工作。特别是,PEAKS Online能够适应目前日益增加规模的蛋白质组学数据吞吐量,实现高通量并行计算。此外,它的易扩展、高性能的设置方式,可以通过改变硬件配置随时对吞吐量和性能进行动态调整。如果拥有合适的计算资源,PEAKS Online 可以加快数据处理的速度,至少比PEAKS Studio快10倍,并且可以处理1000个或更多的样品队列。
在最近的一项可扩展性的测试研究中,我们通过增加计算资源来评估PEAKS Online 的可扩展性。测试数据集包含56个样本,每个样本由12个组分组成。总共有672个3小时的MS run,包括500万次MS1扫描和3000万次MS2扫描。使用标准的PEAKS Online X 32线程license,从数据上传、data refinement,de novo sequencing、PEAKS DB、PEAKS PTM和SPIDER到完成该项目大约需要10天的时间。然而,随着CPU内核的增加,性能呈线性增长。在512 核心数的硬件条件下,花了半天多一点就完成了整个分析。
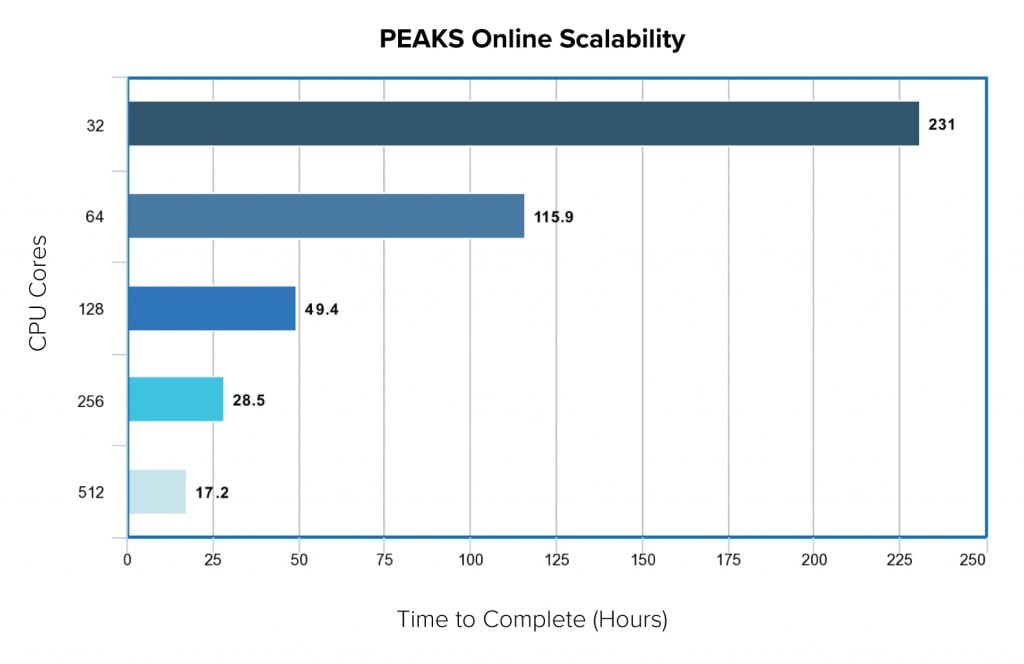
Summary of Dataset Used for Benchmark Analysis
| # of Samples | 56 |
| # of MS/MS Runs (180 mins.run) | 672 |
| # of MS | 5106521 |
| # of MS/MS | 28858408 |
PEAKS Online的license是基于服务器计算资源授权和客户端数量授权而设置的。PEAKS Online的基本配置是5个客户端/64逻辑核心或者线程数。研究人员可以增加计算资源授权和/或用户数量,以满足任何研究小组的需求。可以购买PEAKS Online Server性能和客户端的数量来达成数据处理的理想解决方案。
| # of Clients | # of Cores/Threads |
|---|---|
| 5 | 64 |
| 6 | 128 |
| 7 | 256 |
| 8 | 512 |
*注意,核心数或线程数是针对在PEAKS Online服务器上worker节点可使用的核心数/线程数而言。用户可以在基本配置的基础上,额外订购客户端的数目和授权。PEAKS Q和PEAKS IMS模块也可以额外选购。
总的来说,PEAKS Online 由3个组件组成:数据库节点、主节点(Master)和工作节点(Worker)。这些节点可以部署到不同的计算机,也可以部署在同一台机器上。每个组件都有独特的计算资源使用和硬件需求模式。
PEAKS Online 的数据库节点存储所有应用数据,是所有蛋白质组学数据处理的基础。因为PEAKS Online 是一个可以在多台机器上运行的分布式计算框架,所以PEAKS Online使用流行的分布式数据库系统Cassandra作为主要的数据存储来提供大规模的I/O性能。作为Cassandra集群的一部分,每个数据节点都对内存大小和磁盘I/O速度有很高的要求。
Master节点是PEAKS Online 计算框架的核心,它负责计算任务的调度、分派和同步。尽管它不执行任何数据处理,但它负责基于web的用户界面、加载原始数据和导出结果数据,并将从高性能CPU中获得更多好处。
Worker节点负责实际数据的处理和运算。PEAKS Online 易于扩展,所以worker节点可以配置为使用自定义的CPU线程数量。根据经验,每个worker需要2GB可用内存用于每个计算线程,另外还需要2GB备用内存用于自己的使用。除了用于日志记录的几GB硬盘空间需求之外,工作节点通常对硬盘I/O速度和空间没有要求。
| 64 Thread Licence | 128 Thread Licence | |
|---|---|---|
| Master | 8 线程 ,16 GB内存 | 16 线程, 32 GB 内存 |
| Cassandra | 8 Threads Each, 16 GB Memory Each, SSD (5T+) | 8 线程/每个数据库节点, 16 GB 内存/数据库节点, SSD (5T+) |
| Workers | (16x): 64 Threads Total (4 Threads Per), 160 GB Memory (10 GB Per) GPU Worker: 8 threads, 8G GPU memory, 32G system memory. | (16x): 128 线程 (8 线程/worker), 320 GB 内存 (20 GB Per), GPU Worker: 8 线程, 24G GPU 显存, 32G系统内存. |
| Suggested Configuration | All-in-one 配置方案: INTEL XEON GOLD 6238 处理器 X2 (88 线程,启用超线程) 内存: 256 GB SSD: 500G (系统) SSD: 5T (数据库节点) GPU: Nvidia GTX 1080 or Nvidia Quadro P5000 16GB | 两台机器组建集群,每台配置推荐如下: INTEL XEON GOLD 6238 处理器 X2 (88 线程,启用超线程) 内存: 256 GB SSD: 500G (系统) SSD: 5T (数据库节点) GPU: Nvidia RTX 3090 或 Nvidia Quadro RTX A6000 |
注意:虽然PEAKS Online 最好在具有数十到数百个节点的集群上部署以达到最好的计算效率,但如果您只有有限的计算资源,一台功能强大的工作站/服务器,您仍然可以在all-in-one的模式下使用它。事实上,PEAKS Online的所有组件都可以从同一台机器上启动。但是在这种情况下,您必须确保有足够的计算资源来满足所有组件的需求。数据库对硬盘的读写非常频繁。如果所有东西都安装在同一台机器上,强烈建议为数据库组件单独准备一个固态硬盘的磁盘阵列组。
- Lightbend, Inc. (2011-2019). Akka: Build powerful reactive, concurrent, and distributed applications more easily. https://akka.io/.
- The Apache Software Foundation (2016). Apache Cassandra. http://cassandra.apache.org/.
- Xin, L., Qiao, R., Chen, X. et al. A streamlined platform for analyzing tera-scale DDA and DIA mass spectrometry data enables highly sensitive immunopeptidomics. Nat Commun 13, 3108 (2022). https://doi.org/10.1038/s41467-022-30867-7
资源
- PEAKS Online 11 Brochure
- Xin, L. et al. Towards Real-time Proteomics Data Analysis using PEAKS Online 8.5. ASMS. ThP 461. 7/6/2018.