

众所周知,PEAKS被业界认为是多肽从头测序的金标准。为了进一步推动从头测序技术的发展,BSI率先将深度学习技术引入质谱数据的多肽从头测序中,并报道了其准确性显著提高[1,2]。这项技术的速度和准确性自然驱动了我们将DeepNovo整合到PEAKS的解决方案中。
作为从质谱(MS)数据中发现全新多肽的关键技术,DeepNovo提供了先进的解决方案,并将进一步推动抗体测序和新抗原发现等研究。
功能核心:
- 增强型DeepNovo整合了GraphNovo,一种基于图形的深度学习从头测序,可提高氨基酸和多肽水平上的准确度;
- 利用 GPU 加速从头测序
- 为多肽测序结果提供了无偏 FDR评估
- 在不同样本类型中观察到的基于下一级深度学习的 DeepNovo 多肽测序的改进
在最新的GraphNovo算法的支持下,新的DeepNovo测序算法通过在第一阶段找到最佳路径来指导第二阶段的序列预测,解决了由于片段离子缺失而导致的预测误差累积的问题。

多肽测序的FDR控制
PEAKS12实施了一种新的无偏方法来评估从头肽测序方法,使用目标诱饵光谱来估计其FDR。通过保持诱饵和目标MS2光谱之间的峰值数量相似以及通过确保目标和诱饵峰值的非常相似的m/z和强度分布,从目标MS2频谱创建诱饵MS2光谱。除了基于%ALC(平均局部置信度)评分的传统过滤外,DeepNovo中还提供了从头测序的FDR曲线。
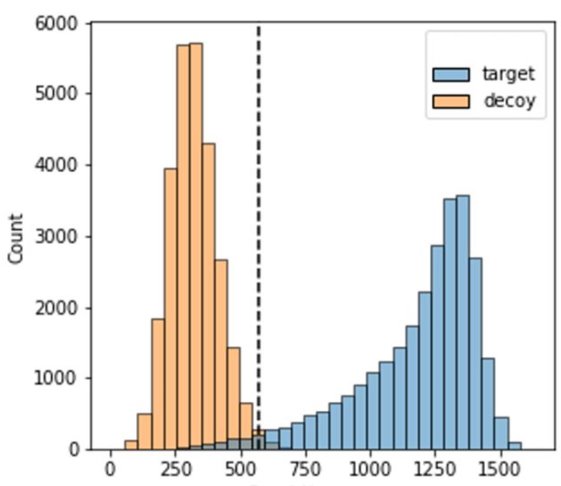
下一级DeepNovo测序,提高了准确性和灵敏度。
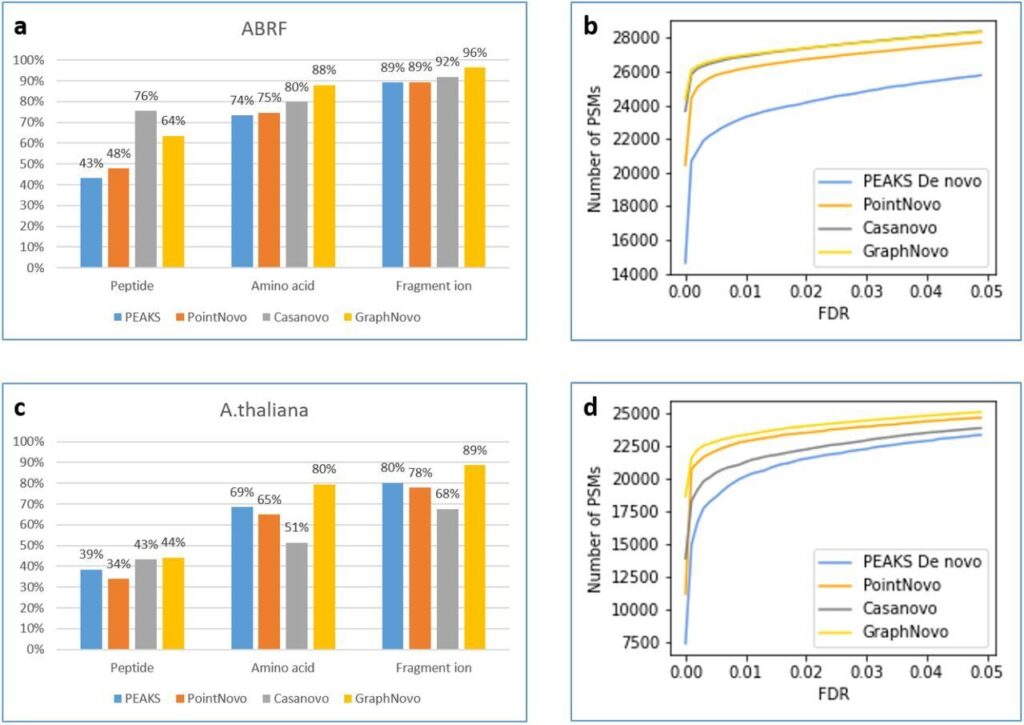
使用来自不同来源(包括人类和HLA数据集)的原始数据,将PEAKS 12最新的GraphNovo测序算法与传统的PEAKS Denovo测序算法以及的基于深度学习的PointNovo和Casanovo算法进行了比较。
对于人类数据集,在1%的FDR阈值下,PEAKS、PointNovo、Casanovo和Graphnovo分别报告了23300、26194、26892和26982个PSM。Graphnovo在肽、氨基酸和片段离子水平上的准确率分别为63%、85%和96%,表现最好。
在HLA数据集中也得到了类似的结果,这使得PEAKS12下一级DeepNovo测序成为新肽发现和免疫肽分析的关键。
参考文献与引用
References
- Tran, N. H., et al. (2017). De novo peptide sequencing by deep learning. Proceedings of the National Academy of Sciences of the United States of America, 114(31), 8247–8252. https://doi.org/10.1073/pnas.1705691114
- Tran, N. H., et al. (2019). Deep learning enables de novo peptide sequencing from data-independent-acquisition mass spectrometry. Nature methods, 16(1), 63–66. https://doi.org/10.1038/s41592-018-0260-3
- Phulphagar KM, et al. (2023). Sensitive, high-throughput HLA-I and HLA-II immunopeptidomics using parallel accumulation-serial fragmentation mass spectrometry. bioRxiv. 532106. doi: 10.1101/2023.03.10.532106
- Tretter C, et al. (2023). Proteogenomic analysis reveals RNA as a source for tumor-agnostic neoantigen identification. Nat Commun. 14(1):4632. doi: 10.1038/s41467-023-39570-7

