de novo Peptide Sequencing
功能特点:
- 与深度神经网络模型结合,DeepNovo用于多肽从头测序,提高氨基酸和多肽水平的精确度
- 高通量、自动化的从头测序
- 精确的算法,提供de novo结果可信度打分,确保氨基酸水平的精确度
- 可以与database search整合,深度挖掘蛋白质组的未知信息
- 支持CID、HCD、ETD/ECD 、EThcD、EAD等多种碎裂模式

de novo Peptide Sequencing
在串联质谱中,质谱通过对肽链碎裂产生的碎片离子检测,进而产生ms/ms谱。根据所采用的碎裂方法,可以产生不同的碎片离子类型。未知多肽的测序是在不需要序列数据库的情况下,从质谱中解析出氨基酸序列。这与另一种流行的肽段鉴定方法–“数据库搜索”形成了鲜明对比,即在给定的数据库中搜索与该谱图正确匹配概率最大(或者错配概率最小)的肽。当没有可参考的序列数据库时,多肽的从头测序是唯一的选择。
业界知名的PEAKS系列软件的核心算法之一就有de novo,可以不依赖于数据库高效地进行全自动的肽段序列预测。这使得PEAKS软件成为鉴定未测序生物中新的肽和蛋白质的首选方法。
DeepNovo
DeepNovo是一种基于深度学习的de novo测序算法,通过连续迭代预测氨基酸,从MS/MS图谱中预测多肽序列。DeepNovo一个接一个预测氨基酸,并计算其得分。氨基酸水平的分数表示特定氨基酸在多肽序列中某个位置出现的概率。然后通过各个氨基酸得分的平均值作为多肽的得分。该工具可以提高氨基酸水平的准确性,提高氨基酸和肽的召回率。
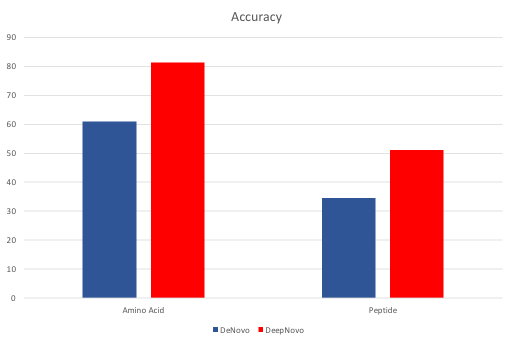
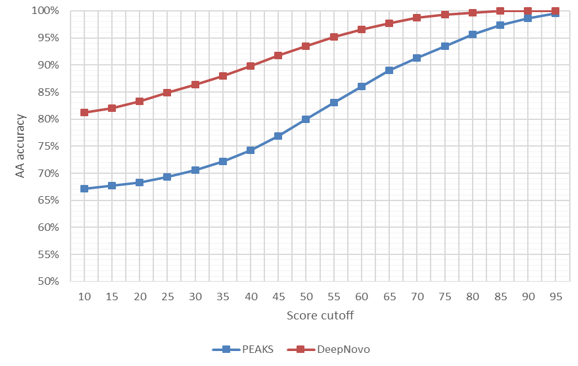
精确度
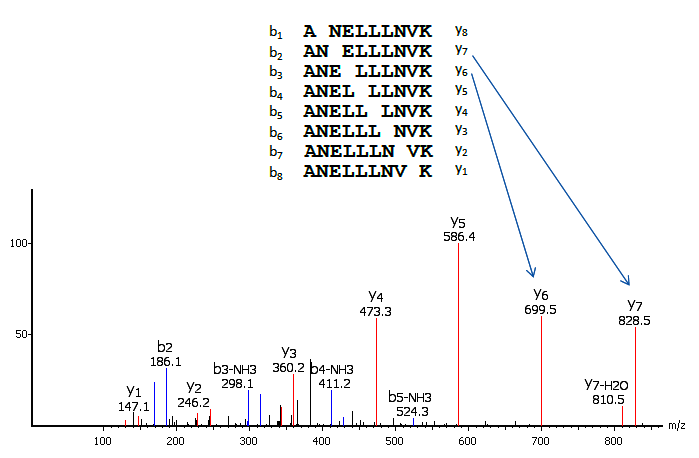
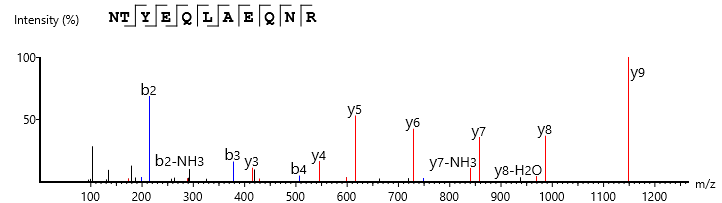
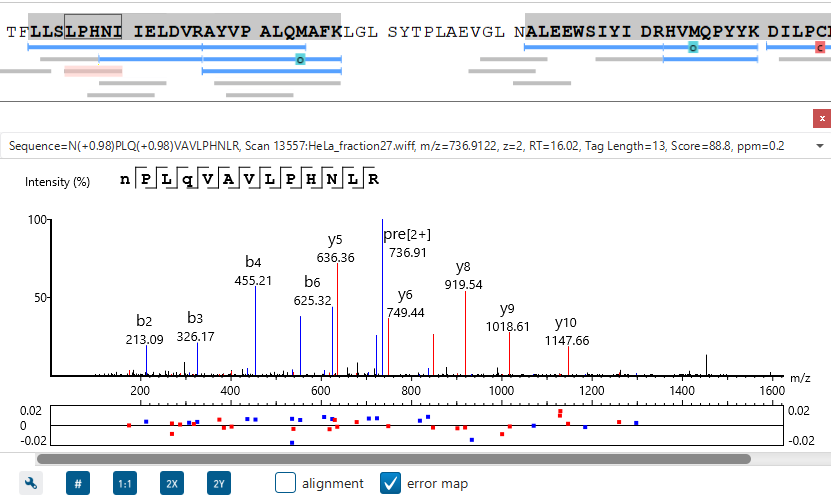
PEAKS使用了全面综合的打分体系,来对于de novo的肽段序列结果的准确性进行打分评估。PEAKS的独特之处在于local confidence(LC score)评分–评估结果中肽段每个氨基酸分配的可能性。local confidence score将测序的准确度聚焦到氨基酸水平。在图中, TYEQLAEQNR 是通过了可靠性阈值的序列标签。

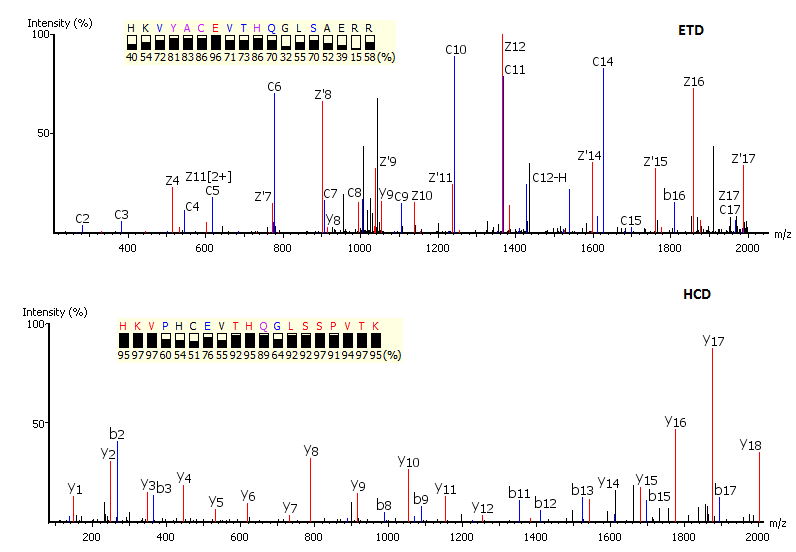
互补的碎裂离子
碎裂方式互补配对的谱图使用:de novo从头测序可以使用由不同的碎裂方式(例如ETD/HCD)产生的成对的谱图。每个互补的谱图产生置信度de novo序列标签用来重构一个肽序列,并对两个谱图进行优化。
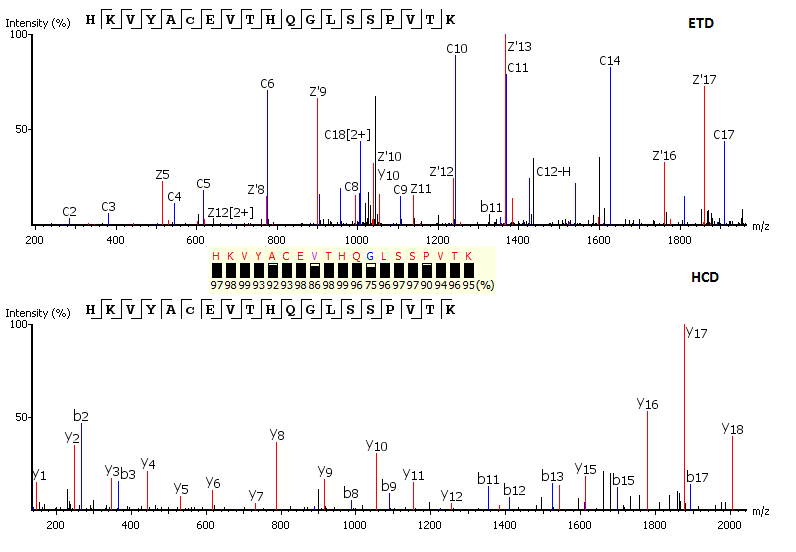
与Database Search整合
PEAKS的独特之处在于能够将多肽de novo测序结果与数据库搜索结果相结合。多肽de novo序列与蛋白质数据库条目比对,以提供关于PTM、突变、同源多肽和全新肽段的附加信息
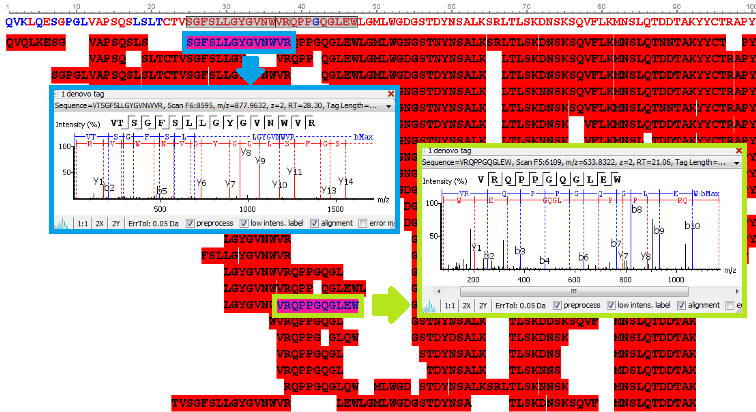
从多肽从头测序到蛋白质测序
蛋白质的序列可以由多肽的de novo测序结果中获得。拥有直接的谱图碎片离子证据的可靠的de novo多肽序列标签,可以用来组装为蛋白质序列。PEAKS de novo应用到蛋白质测序的最佳实例就是在抗体测序方面的软件PEAKS AB–该软件可依据质谱二级谱图所提供的谱峰信息全自动对抗体进行蛋白水平de novo从头测序及序列拼接的工作,高效自动化地代替人工拼接,从而获得目标抗体的全序列,为研发工作带来更大的便利和更好的准确性。
Reference
- Tran NH, Qiao R, Xin L, Chen X, Liu C, Zhang X, Shan B, Ghodsi A, Li M. Deep learning enables de novo peptide sequencing from data-independent-acquisition mass spectrometry. Nature Methods. 16(1), 63-66. 20/12/2018.
- Tran NH, Zhang X, Xin L, Shan B, Li M. De novo peptide sequencing by deep learning. Proceedings of the National Academy of Sciences of the United States of America. 114(29). 18/7/2017.
- Tran NH, Rahman MZ, He L, Xin L, Shan B, Li M. Complete De Novo Assembly of Monoclonal Antibody Sequences Scientific Reports. 6(31730). 26/08/2016.
- Qiao R ,Tran NH ,Xin L ,Chen X , Li M , Shan B, and Ghodsi A, Computationally instrument-resolutionindependent de novo peptide sequencing for high-resolution devices, Nat. Mach. Intell, 3, 420–425 (2021). doi:10.1038/s42256-021-00304-3

