
首次运行PEAKS时,License安装向导页面将自动出现。有以下三种选项:
- 试用或购入的License密钥激活PEAKS:如果您有License密钥,请选择此选项。在“输入许可证密钥”弹出窗口中输入密钥,PEAKS将重新启动。
- 注册以免费获取试用版License密钥:如果您想免费获取15天试用License密钥,请选择此选项。
- 用PEAKS作为浏览器:此选项仅可用于查看已分析好的PEAKS项目。
- 单击License安装向导页面右下角的“手动激活PEAKS”按钮。
- 输入请求的信息,单击下一步。
- 如果您想注册一个试用License ,您可以使用上面的“如何安装PEAKS Studio?”问题中描述的过程来获取License密钥。
- 保存License请求文件,并将其传输至已链接互联网的计算机上。
- 转至 http://www.bioinfor.com/lcs20/index.jsp
- 按照试用申请页面上要求提交License密钥申请,您将在电子邮件收件箱中收到License密钥。
- 将License密钥传输到需要安装PEAKS的计算机上,并将其粘贴到向导的“important licence”步骤中提供的文本框中,单击下一步,继续执行后,PEAKS会重新启动并完全正常运行。
在本页中, 您将看到 PEAKS Studio和PEAKS Online的桌面版和服务器版的推荐配置。
PEAKS可以。 请查看我们的 data types 部分,来了解如何从您的仪器加载数据。
- 在首选项中,选择“Search Engine”,然后选择“Mascot Settings”。
- 您需要知道 Mascot 服务器的 IP 地址或名称以及 Mascot 版本。
- 在‘Hostname (or IP address):’文本框中输入此信息。通常只需输入没有引号的“//mascotserver”。
- 输入‘Port:’编号,默认值为80。
- 进入虚拟目录,默认为’mascot’。
- 在‘Version’下拉菜单中选择您的版本号。
- 如果需要使用用户名和密码登录,可以在版本号下方的框中输入它们。
- 选中“Save Password”框,这样您就不必每次都输入密码。
- 单击“Apply”按钮以保存您所做的任何更改。
*如果 Mascot 已安装在本地计算机上,请在Hostname (or IP address) 字段中键入“localhost”。例如,您的连接显示为“http://mascotserver/mascot”
- 从 Sequest 导出 pepXML 结果报告。
- 设置 inChorus搜索时,选择‘Sequest’下拉列表 >’Import’。
- 选择在Sequest 搜索中使用的数据库。确保您在 PEAKS 中配置了相同的数据库。
- 使用浏览按钮查找导出的 pepXML 文件。
- 单击“OK”以将Sequest的结果导入到PEAKS inChorus的运行中
答案是肯定的。当您上传具有混合碎裂方式的数据时,请选择“混合 CID/HCD/ETD”。PEAKS将根据给定的数据自动检测每次扫描的碎片类型。
如果您是第一次使用PEAKS,请访问页面 supported data file formats。这将解决您99%的相关问题。对于从旧版本更新到最新版本的 PEAKS 的用户,请参考以下方案:
- Thermo原始文件:为了能在PEAKS中读取Thermo 原始文件,用户必须首先卸载32位版本的Thermo MSFileReader,该版本可用于PEAKS 6或更早的版本。然后单独安装64位版本的Thermo MSFileReader,或者从PEAKS中的配置向导中安装。
- Agilent原始文件:为了能在PEAKS中读取安捷伦原始文件,用户必须首先卸载 32 位版本的安捷伦 MassHunter 数据访问组件 (MHDAC)。然后单独安装 64 位版本,或在管理员权限下运行 C:\PEAKSStudio#\MHDAC_x64 目录下的 RegisterMassHunterDataAccess.bat。
- Waters原始文件:PEAKS支持直接读取Waters原始文件,前提是配置了Masslynx。
这个问题通常与PEAKS中使用的第三方工具有关。每当更新第三方工具时,关联的文件夹也必须更新。通过从用户的PEAKS安装目录中删除这些文件夹(例如C:\\Users\[用户名]\PEAKS_8.5\),PEAKS将在重新启动时自动重新生成这些文件夹的新副本(最新版本)。以下操作是解决此问题的正确方法:
- 关闭PEAKS
- 进入 PEAKS 目录(例如:C:\\Users\[username]\PEAKS_8.5\)
- 删除文件夹:“derbyserver”、“defaultTaskDB”和“defaultProject”
- 重新启动PEAKS并尝试重新加载数据
这个问题通常与PEAKS中使用的第三方工具有关。每当更新第三方工具时,关联的文件夹也必须更新。通过从用户的PEAKS安装目录中删除这些文件夹(例如C:\\Users\[用户名]\PEAKS_8.5\),PEAKS将在重新启动时自动重新生成这些文件夹的新副本(最新版本)。以下操作是解决此问题的正确方法:
- 关闭PEAKS
- 进入 PEAKS 目录(例如:C:\\Users\[username]\PEAKS_8.5\)
- 删除文件夹:“derbyserver”、“defaultTaskDB”和“defaultProject”
- 重新启动PEAKS并尝试重新加载数据
- Local Confidence 指的是de novo peptide中整合成这个多肽的每一个氨基酸被分配在这个位置的可信度。它用百分比来表示。
- Total Local Confidence (TLC) 是肽序列中每个氨基酸的 local confidence scores(0到1)的总和。
- Average Local Confidence (ALC) 是TLC的平均值,也是TLC除以肽序列的氨基酸数量得到的值。
通常,肽的结果中55%以上是好的。但这并不意味着整个序列是正确的序列。这是因为在从头测序中开始和结束部分的光谱更难解释,由于这些片段的离子质量高或低。这就是为什么我们给结果设置了不同的颜色,以便您可以看到序列中高置信度的部分。
您完全可以从denovo序列标签末端中去除低评分氨基酸。为此,请点击分数阈值滑块按钮(denovo结果上方的绿色,黄色,和红色符号)。使用这个滑块来设置一个分数阈值,任何低于这个阈值的氨基酸都将缩减为其道尔顿质量。当您导出标签结果,表格的最后一列导出的结果将遵循这个置信度阈值。
最基本的:结果的质量。与其他的软件相比,PEAKS能始终如一地提供更准确的肽序列和更好的置信度。这是PEAKS全局优化算法和复杂评分模式的结果。
-10lgP 分数只是一个递增的 p 值。例如,p 值 1% 等于 -10lgP 为 20。对于给定的分数 x,其相应的 P 值是错误标识分数大于或等于 x 的概率。有关评分系统的更多信息,请参阅 PEAKS DB Score Tutorial.
PEAKS仅要求数据采取FASTA格式,并且标头应仅包含标准ASCII字符。
很可能是因为PEAKS没有找到数据库。在PEAKS中,先找到配置(Configuration),然后数据库(Database)。确保您在下拉菜单中正确的FASTA数据库类型,并在路径文本框中选择了正确的数据库路径。
对于具有中性损失质量数的PTM,PEAKS将搜索修饰氨基酸的同位素质量数以及中性损失质量数。PEAKS并不需要中性损失质量数的存在来鉴定修饰的氨基酸。但如果它存在,确实会有所帮助。
详情请参阅PEAKS DB Score Tutorial 。
- 以Mascot为例,在首选项中,转到“Search Engine”,然后转到“Mascot Settings”。
- 您需要知道 Mascot 服务器的 IP 地址或名称以及 Mascot 版本。
- 在“Hostname (or IP address):”文本框中输入此信息。通常这只是没有引号的“//mascotserver”。
- 输入“Port:”编号,默认值为 80。
- 进入虚拟目录,默认为’/mascot’。
- 在“Version”下拉菜单中选择您的版本号。
- 如果需要使用用户名和密码登录,可以在版本号下方的部分中输入它们。
- 选中“Save Password”框,这样您就不必每次都输入密码。
- 单击“Apply”按钮以保存您所做的任何更改。
*如果 Mascot 已安装在本地计算机上,请在Hostname (or IP address) 字段中键入“localhost”。例如,如果您的连接显示为“http://mascotserver/mascot”
肽百分比分数反映了考虑到所有使用的搜索引擎的肽谱匹配的质量。分数是根据 Peptide-Prophet中使用的经验计算得来的。蛋白质百分比分数是根据肽百分比分数计算得出的。肽百分比分数是每种蛋白质的加权和合并。然后将PeptideProphet方法应用于整套蛋白质,以获得最终的蛋白质概率分数。
“-1”表示发现了一个比率,但它并不来自任何高分肽。如果要显示该比率,可以在运行参数设置中提高肽的评分阈值。
最可能的原因是用户必须至少有两个样本,并且每个样本中的片段数量相同。
当存在潜在的内存问题时,最好检查PEAKS的性能配置,以确保计算机资源分配正确。设置性能配置时的一般规则.exe(默认情况下位于 C:\PEAKSStudioXplus 文件夹中)是仅将计算机物理资源的 75-80% 分配给 PEAKS。
推荐的计算机资源和性能配置
| Computer Resources | JVM Heap Size (MB) | Computing Nodes to Start | |
|---|---|---|---|
| 16-thread desktop licence | 64 GB RAM, 20 logical processors |
48000 | 16 |
| 32-thread workstation licence | 128 GB RAM, 40 logical processors |
96000 | 32 |
以上是建议的计算机资源和性能配置,以全面优化PEAKS许可证。如果需要手动配置,请确定可以为峰值分配的线程和 RAM 数量。对于分配给 PEAKS 的每个线程,需要 3 GB 的内存。
- 最好将所有文件(示例文件、项目和数据库)存储在本地驱动器中。
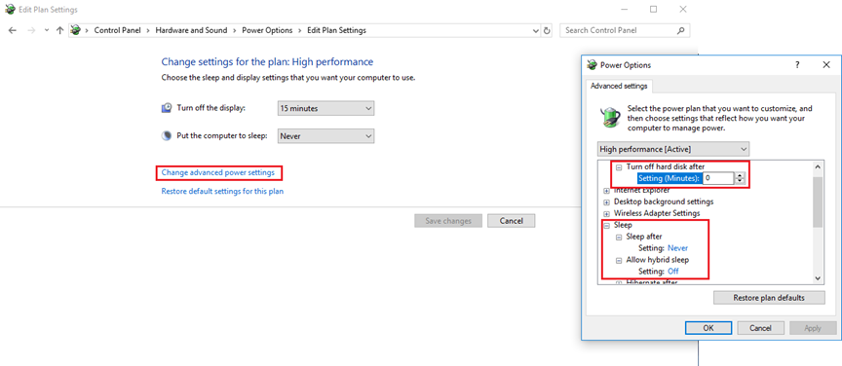
- 确保电源设置设置为永不休眠
o 转到开始菜单,然后通过键入“控制面板”打开控制面板
o 在右上角的搜索栏中,键入“电源”并打开“更改电源设置”
o 切换到高性能选项
o 另外,选择“更改计划设置”,然后选择“更改高级电源设置”
o 找到“关闭硬盘电源”并将其切换为0,这意味着硬盘电源不会关闭
o 同时将所有睡眠选项设置为“从不”
- 所有类型的数据文件都没有加载,进度条卡在 0%。
这可能与集成到PEAKS中的第三方工具(称为derbyServer)有关。由于某种原因,与文件夹关联的读/写权限可能已更改。如果使用以下方法重置文件夹,则应防止此问题:- 登录使用峰值的帐户时,请确保关闭峰值
- 转到 C:\Users[您的用户名]\PEAKS_10.5 并删除 defaultproject、defaultTaskDB 和 derbyserver
- 当您重新启动 PEAKS 时,将使用默认权限以默认格式重新创建这些文件夹,并且 PEAKS 应该运行平稳。
- 添加了多个文件,但有些文件未成功加载(绿色¾图标未填充)或数据加载进度条上升到 99% 然后失败。这更可能是内存问题,通常发生在较大的原始文件上。将raw_file_loader_heap_size=512从 C:\Users[用户名]\PEAKS_10.0\PeaksInternal.properties 文件中增加到1024(如果有足够的计算机资源,则增加到2048)。
确保数据加载和数据细化成功完成的一般提示:
确保 MS1 和 MS2 扫描编号不为0。要检查这一点,请单击示例文件并检查“Properties”选项卡。
- 检查 C盘和存储项目的本地驱动器中是否有足够的可用磁盘。大型项目可能需要 C盘中最多50 GB 的可用磁盘,并且硬盘驱动器中的内存需要是当前项目大小的两倍。
- 检查参数中是否有可能导致此问题的内容。通常,修饰列表必须至少保持最小(如果可能的话,大约 3-5 个)以减少搜索空间。其他修饰可以在PEAKS PTM搜索中使用“高级设置”指定。
- 如果有足够的计算机资源,则增加derby内存。来到 C:\Users[用户名]\PEAKS_10.5\PeaksInternal.properties,然后在记事本中编辑该文件。将 java_vmArg_derby=-Xmx1024m 增加到 2048 (2 GB)。
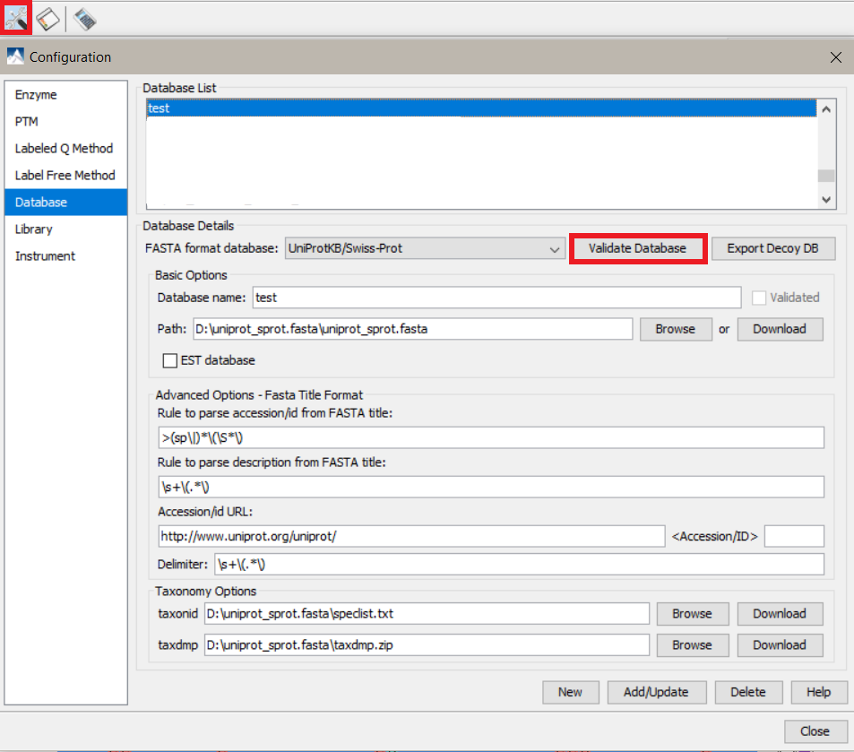
- 检查PEAKS是否能够正确读取fasta文件,或者是否有任何无效内容,方法是转到 Preferences > Database ,然后单击“Validate Database”选项。
- 减小一个批次的大小以防止内存不足错误。
- 转到 C:\PEAKSStudioXplus\algorithmpara 并在记事本中打开 batchSizeConfig.xml
- 然后,将 5000 更改为 2500
- 这将减少通过 PEAKS DB 运行的批处理的大小
- 然后,将 1.3 更改为 2.0
- 这将增加分配给搜索每个谱的内存量
在某些情况下,PEAKS搜索已完成,但没有肽,或者鉴定出很少的肽。如果指定的质量误差容差(mass error tolerances)对数据严格,则可能会发生这种情况。以下是每种仪器的建议质量误差容差。
建议的仪器 Mass Error Tolerances
| Instrument | Parent Mass Error Tolerance | Fragment Mass Error Tolerance |
| Orbi-Trap | 15.0 ppm | 0.5 Da |
| Orbi-Orbi | 15.0 ppm | 0.02 Da |
| Triple-TOF | 15.0 ppm | 0.1 Da |
| QTOF | 0.1 Da | 0.1 Da |
| TIMS-TOF | 15.0 ppm | 0.1 Da |
| TOF-TOF | 0.1 Da | 0.1 Da |
| Ion Trap | 0.5 Da | 0.5 Da |
| Linear Ion Trap | 0.5 Da | 0.5 Da |
以上是每种仪器的默认质量误差容差。这些可以通过参考图2b PEAKS肽评分与母离子质量数误差的散点图,和图6过滤结果中肽谱匹配(PSM)的母离子质量误差来根据需要进行调整。
其他产品 & 服务
有关产品和服务的其他问题,请联系 support。

