
PEAKS AB 软件的自动化流程以及可编辑的人工核验模式可以进一步提高测序的准确性。它还提供了一个更加方便和用户友好的工作流程,用于抗体测序分析,并且可以为用户提供新的定制功能。
单克隆抗体生化分析、医学研究、诊断和治疗中都有非常广泛的应用。抗体(Ab)蛋白的序列信息编码了它的结构域功能的重要信息。而当cDNA或者杂交瘤细胞无法获得时,就需要对蛋白进行de novo测序。
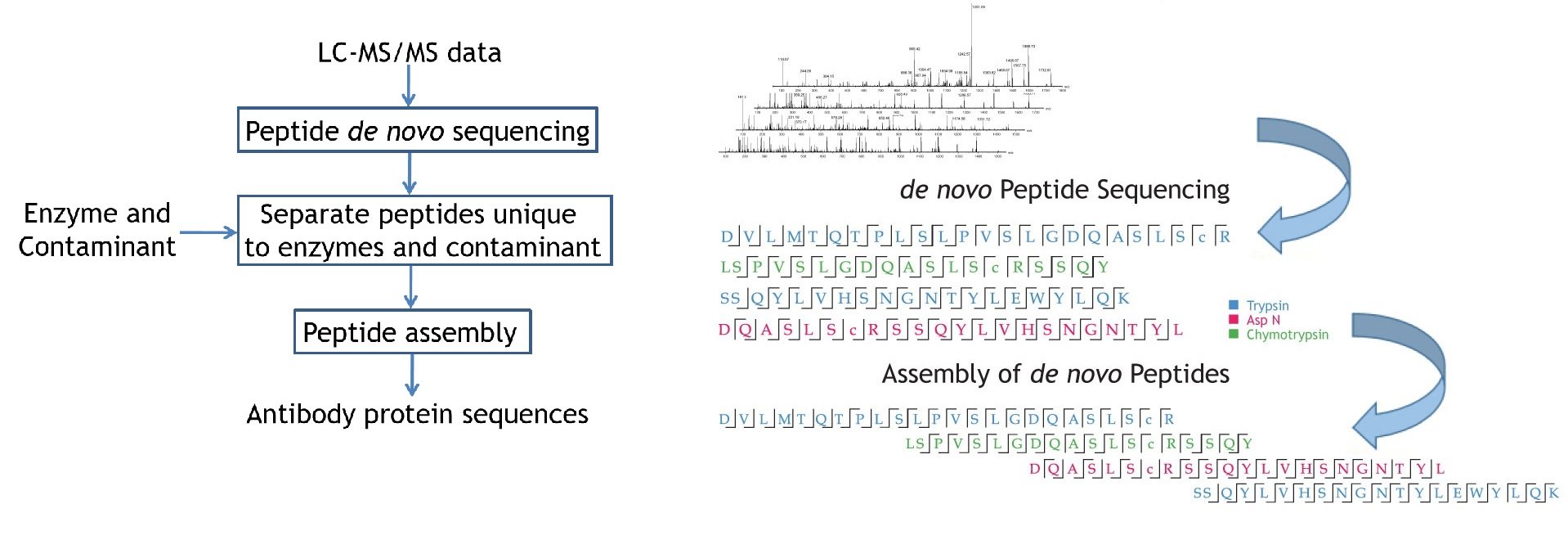
PEAKS AB软件是一个非常强大的基于高精度液相-质谱数据集,全自动对抗体进行de novo 从头测序,并对其修饰和序列突变分析进行表征的分析平台。PEAKS AB软件同样也支持对研究发现和质量控制为分析目标的翻译后修饰(PTMs)和序列突变的表征。软件能够从质谱原始谱图数据,首先提供肽段de novo测序结果,并将其中可靠的序列标签组装为完整抗体蛋白序列。
软件概览
从抗体的肽段de novo测序结果得到抗体蛋白序列。 这些可靠的从头测序序列标签是来源于碎片离子的直接证据,进而组装为抗体序列。可通过点击肽段查看谱图,并且直接在谱图上标注碎片离子对应的每一个氨基酸。
de novo View
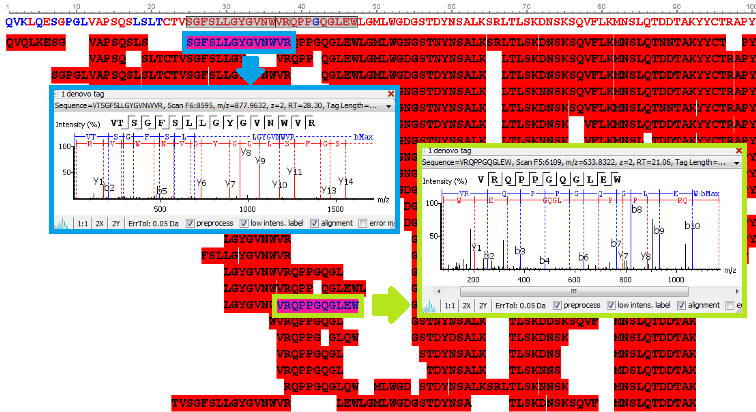
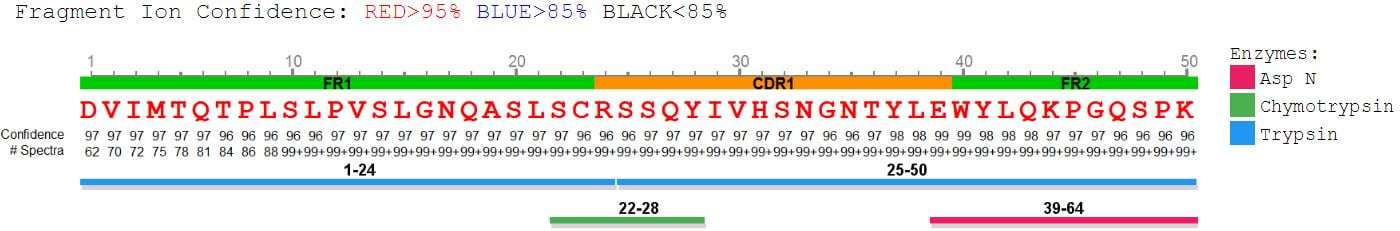
PEAKS AB: 提供综合的肽图结果,包括:
1. 全序列信息整合视图
2.显示每一个氨基酸来自碎片离子的直接 证据
3.翻译后修饰及位点信息(PTMs)
4.氨基酸替换(序列突变确证)
Peptide Mapping View

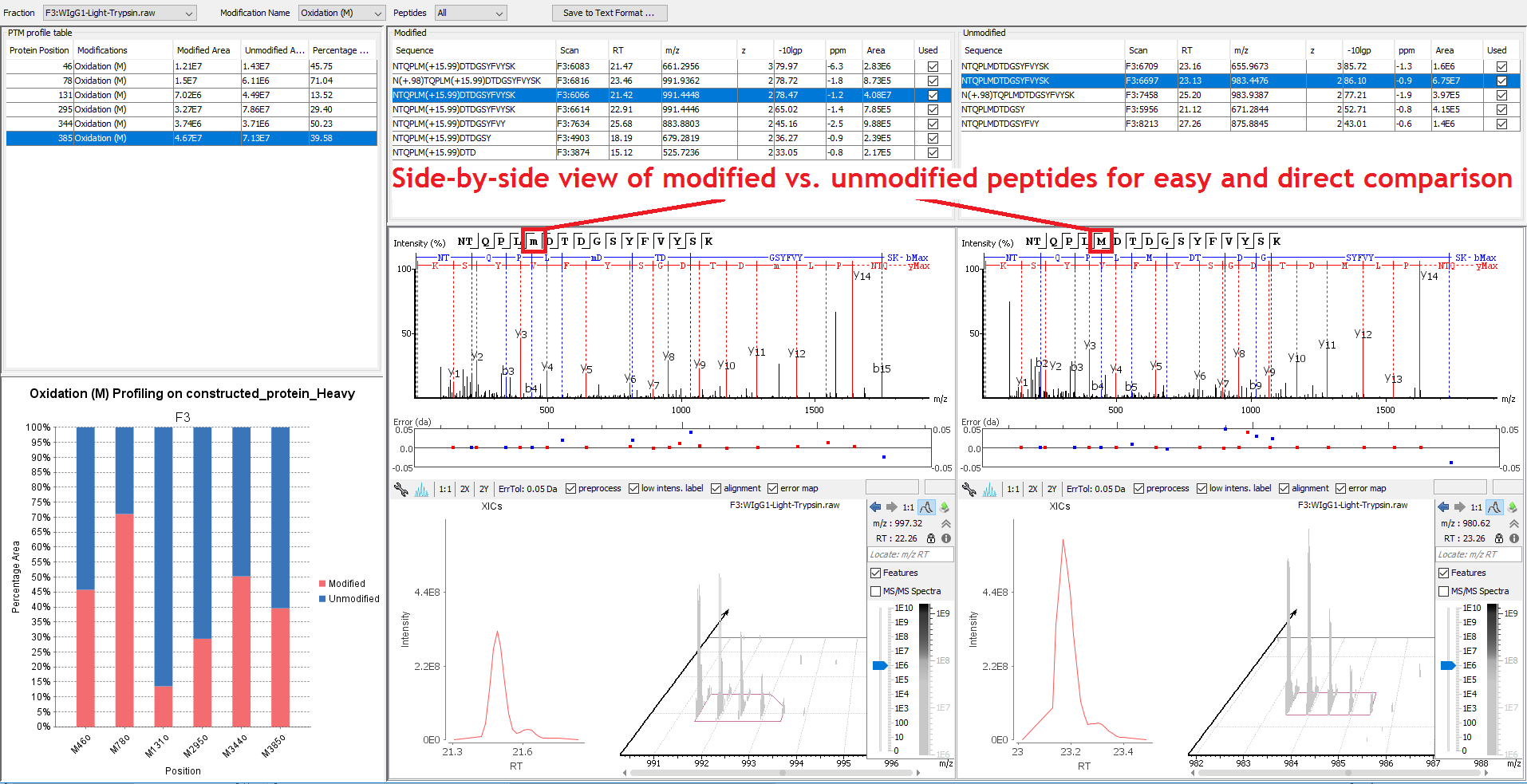
对于含有修饰位点的每一条序列,在定量结果视图中,都列出了它们的修饰形式和未修饰形式,及其相应的色谱图,提供基于XIC的定量信息。
翻译后修饰(PTMs) 分析
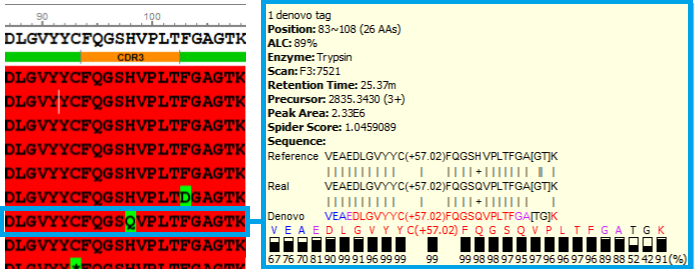
序列突变视图对于可能是由互补DNA的转录错误产生的氨基酸变异和/或截短的结果部分予以高亮显示。
序列变异分析视图
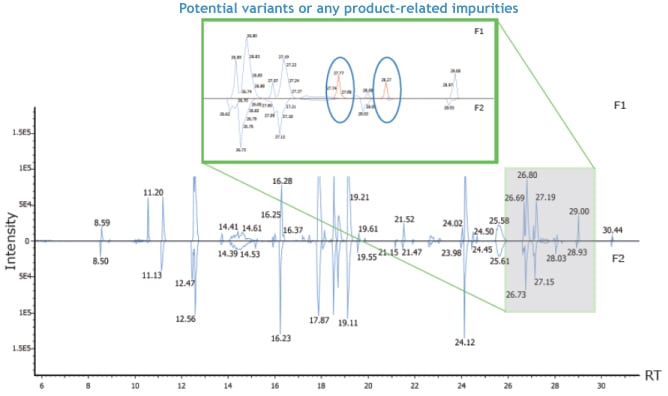
在强化的peptide mapping功能中,PEAKS AB软件可以比较、注释不同样品的色谱图。软件中阐明可能发生的序列突变,以便于产品质量控制或任何其他相关的抗体研究。
如下图所示,带有注释的色谱图中蓝色的峰表示信号映射到来自参考蛋白质的肽段,而红色的峰表示序列突变或任意与产品相关的杂质。
肽图
PEAKS AB不仅能支持EThcD碎裂模型,而且还能将不同碎裂类型的同一个母离子的MS2谱图结合起来进行从头测序,从而得到更可靠的从头测序结果。
EThcD数据分析

算法
PEAKS AB软件可以利用正交设计的多酶联合酶解的抗体蛋白,通过高分辨率LC-MS/MS获得的数据集,直接对抗体蛋白进行序列分析。该方法首先进行肽段的de novo sequencing,并将可信度高的序列标签自动组装成抗体序列。
报告生成
- 抗体样品描述
- 缩略词说明
- 实验流程
- 抗体轻/重链信息
氨基酸序列
通过肽谱匹配水平0.1% FDR质控的peptide mapping
可变区典型肽段的选择
区分Ile/Leu
References
- Shan, B. and Xin, L. Integrating de novo Sequencing and Database Search for Monoclonal Antibody Sequencing. J Biomol Tech. 24(Suppl). S62–S63. 1/5/2013.
- Tran, N.H. et al. Complete De Novo Assembly of Monoclonal Antibody Sequences. Scientific Reports. 6:31730. 26/08/2016.


